Las cistatinas son un grupo de proteínas identificadas en vertebrados, invertebrados, plantas y algunos microorganismos, que inhiben cisteín-proteasas. Las cistatinas de plantas (fitocistatinas) constituyen una familia formada por más de 80 miembros descritos tanto en mono- como en di-cotiledóneas, que se agrupan en una rama independiente del árbol evolutivo de esta superfamilia de proteínas. Se caracterizan por presentar, además de la secuencia conservada LARFAV exclusiva de vegetales, tres motivos comunes a todas las cistatinas implicados en la interacción enzima-inhibidor: i) el sitio reactivo QxVxG; ii) una o dos glicinas en el extremo amino terminal de la proteína y iii) un triptófano en la segunda parte de la molécula (Figura 1).
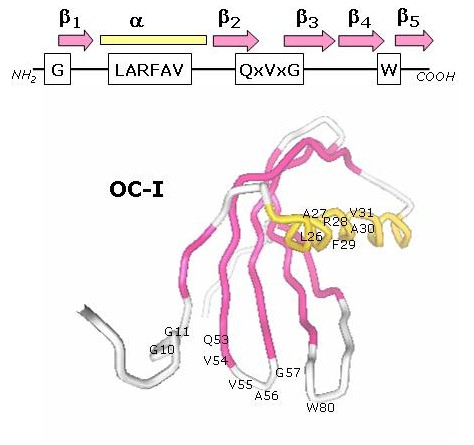 |
Figura 1. Esquema y estructura tridimensional de la orizacistatina I de arroz (OC-I) indicando |
Las fitocistatinas son inhibidores no competitivos que inhiben de forma específica peptidasas C1A de la familia de la papaína (peptidasas C1A; MEROPS peptidase database, http:merops.sanger.ac.uk). La afinidad de cada fitocistatina por las distintas peptidasas es específica y diferencial como consecuencia de un proceso evolutivo cuya finalidad es la ampliación del espectro de inhibición (1). Además, presentan puntos isoeléctricos neutros o ácidos, carecen de puentes disulfuro, no sufren procesos post-traduccionales de glicosilación y son estables a variaciones de temperatura y pH. La mayoría posee un peso molecular entre 11-16 kDa y un solo dominio inhibidor, aunque se han descrito algunos miembros que presentan una extensión carboxilo terminal que las confiere un tamaño molecular de ~ 23 kDa (2,3). Recientemente se ha demostrado que los genes que codifican fitocistatinas de bajo peso molecular carecen de intrones o poseen solo uno localizado entre el motivo LARFAV y el centro reactivo de su región codificante (ORF), mientras que los genes que codifican proteínas de ~ 23kDa presentan sus ORFs interrumpidos por tres intrones (3, 4). El significado funcional y/o evolutivo de los intrones es aún desconocido. Además, existe otro grupo independiente de fitocistatinas, denominadas multicistatinas de ~ 87 kDa, que están formadas por la repetición de 8 dominios inhibidores (5, 6).
A pesar del elevado número de fitocistatinas descritas, solo se dispone de datos cristalográficos precisos de la orizacistatina I de arroz (OC-I), cuya estructura tridimensional ha sido determinada por espectroscopía de resonancia magnética nuclear. Dentro de esta estructura aparece una a -hélice y cinco láminas b antiparalelas (Figura 1) (7). A partir de esta estructura y mediante métodos bioinformáticos se han modelizado otras fitocistatinas (8, 9). La predicción de la estructura tridimensional de estas proteínas, unida al conocimiento de la estructura 3D de la interacción entre estefina B y papaína, y entre estefina A y catepsina H (10, 11), ha permitido esclarecer la posición de los motivos implicados en la interacción proteasa-fitocistatina. Estos motivos se localizan en el extremo N-terminal (G), y en los bucles entre las láminas b 2 y b 3 (QxVxG) y b 4 y b 5 (W).
Desde la caracterización de la OC-I, el primer inhibidor de cisteín proteasas de plantas descrito en 1987 (12), el número de fitocistatinas identificadas se ha incrementado de forma notable. Un ejemplo claro lo constituyen las cistatinas de cereales, de las que ya se habían descrito en el año 2002, cinco miembros en trigo (13, 14), tres en maíz (15), dos en arroz (12, 16), una en cebada y otra en sorgo (17, 18).
La secuenciación del genoma completo de arabidopsis y arroz y la existencia de numerosas colecciones de ESTs y BACs de otras especies han permitido que en los últimos años se identificaran las primeras familias génicas completas de fitocistatinas en varias especies. Así, Martinez et al. (4, 19) publicaron la anotación de doce cistatinas en arroz, siete en arabidopsis y trece en cebada y propusieron modelos evolutivos basados en la relación filogenética entre esas especies. Simultáneamente, Massonneau et al. (20) identificaron diez cistatinas de maíz y analizaron sus patrones de expresión en respuesta a estreses abióticos. Asimismo, se han identificado diez cistatinas en kiwi, siete en manzano y 25 en caña de azúcar, aunque la caracterización molecular y/o funcional de las mismas aún no se ha completado (1, 21).
Los genes que codifican las distintas cistatinas dentro de la misma especie muestran patrones de expresión diferenciales y capacidades inhibitorias específicas (20). Esta diversidad funcional se puede justificar como el resultado de un proceso evolutivo paralelo al aumento del número, localización y/o función de las cisteín-proteasas de la planta, dianas de estos inhibidores.
Las fitocistatinas se han purificado principalmente de semillas (3, 12, 17, 22), aunque también se han descrito en hojas (23), raíces (17, 24), tubérculos (5), tallos (23), polen (24), botones florales (2) y frutos (25). Incluso se ha descrito una cistatina extracelular insoluble en cultivos celulares de zanahoria (26).
Su localización subcelular no está muy estudiada, aunque se ha descrito una multicistatina de patata en el citoplasma de las células del tubérculo, donde forma cristales, y en las vacuolas de las células del tallo donde se acumula en repuesta a ácido jasmónico (27). Recientemente, mediante técnicas inmunológicas se ha detectado la presencia de cristales de una multicistatina de tomate en el citosol de las células de mesófilo de hoja tratadas con ácido jasmónico (28). No obstante, la mayoría de los genes que codifican fitocistatinas poseen un péptido señal en su extremo amino que implica una localización no citoplásmica de la proteína (4).
A las fitocistatinas se les ha atribuido esencialmente un papel endógeno relacionado con el “turn-over” proteico y la muerte celular programada debido a su capacidad de inhibir cisteín-proteasas de la planta, y un papel de defensa basado en su actividad inhibitoria frente a cisteín-proteasas heterólogas.
La función endógena está poco estudiada, aunque son varias las fitocistatinas relacionadas con la degradación de las proteínas de reserva de las semillas. Se han demostrado la capacidad de la OC-I de arroz y la CC-I de maíz de inhibir in vitro las cisteín-proteasas de las propias semillas de arroz y maíz encargadas de hidrolizar las glutelinas y zeinas, respectivamente (29, 30). También se ha determinado el papel regulador que ejercen las cistatinas WC5 de trigo y HvCPI-1 de cebada, sobre la expresión de ciertas cisteín-proteasas durante el desarrollo y germinación de la semillas (14, 31).
Asimismo, se ha demostrado que las fitocistatinas son capaces de inhibir la actividad de proteasas endógenas relacionadas con otros procesos fisiológicos, lo que apoya su papel como reguladores de la proteolisis. Este es el caso de la cistatina de papaya que inhibe in vitro caricaína, una cisteín-proteasa endógena (32).
Existen varias referencias bibliográficas que describen la implicación de cisteín-proteasas y sus inhibidores las cistatinas en procesos relacionados con la muerte celular programada (MCP) en plantas. Solomon et al. (33) comprobaron que la expresión ectópica de una cistatina de soja inhibe la actividad de cisteín-proteasas endógenas y bloquea la respuesta de MCP inducida por una infección de una cepa avirulenta de Pseudomonas o por estrés oxidativo. Igualmente, la sobre-expresión de la fitocistatina AtCYS1 de arabidopsis, en la propia especie, bloquea la MCP activada por herida, patógenos avirulentos y óxido nítrico (8). Recientemente se ha demostrado que una cistatina de Celosia cristata es capaz de conferir resistencia frente al virus del mosaico del tabaco (TMV) en plantas de Nicotina glutinosa , al bloquear las cisteín-proteasas que se activan durante el proceso de MCP en respuesta a la infección vírica (34).
La implicación de las fitocistatinas como proteínas de defensa está mucho más estudiada y avalada por:
Ensayos de inhibición in vitro con fitocistatinas purificadas directamente de la planta o como proteínas de fusión expresadas en Escherichia coli o Pichia pastoris, han demostrado que son capaces de inhibir las cisteín-proteasas presentes en los tractos salivares y digestivos de varios organismos fitófagos. Concretamente, coleópteros, ácaros y nemátodos poseen cisteín-proteasas como los enzimas mayoritarios responsables de hidrolizar las proteínas de su dieta. La actividad proteasa de extractos crudos preparados a partir de estos organismos es bloqueada por las fitocistatinas, de forma que estas actúan como proteínas antimetabólicas que interfieren el proceso de digestión. Las plantas han convertido estas características en una estrategia de defensa y la han adoptado de forma extensiva.
Entre los numerosos ejemplos de inhibición in vitro publicados es de destacar el caso de las cistatinas de arroz, soja y cebada, que purificadas a partir de cultivos de E. coli recombinantes, inhiben la actividad digestiva de importantes insectos plaga como el escarabajo de la patata ( Leptinotarsa decemlineata ) o la crisomela del maíz ( Diabrotica virgifera ), coleópteros que provocan graves pérdidas en las cosechas (35, 36). Recientemente, se ha desarrollado una estrategia nueva para llevar a cabo los bioensayos, incorporando dosis controladas de una cistatina de Vigna unguiculata purificada como proteína de fusión a partir de cultivos de E. coli , en semillas artificiales que se han utilizado para alimentar larvas del coleóptero Zabrotes subfasciatus . Una reducción drástica del peso de las larvas y un aumento de su mortalidad han demostrado los efectos deletéreos de esta cistatina (37).
Pernas et al. (38) demostraron que la actividad cisteín-proteasa de extractos proteicos preparados del ácaro del polvo, Dermatophagoides faringe, era inhibida por una cistatina de castaño purificada de cultivos de E. coli . Recientemente, Carrillo et al. (datos no publicados) han comprobado que las cistatinas de cebada inhiben in vitro los enzimas digestivos de la araña roja ( Tetranychus urticae ), un ácaro fitófago .
Se ha estudiado también el papel de las fitocistatinas como inhibidores de cisteín-proteasas de virus. Aoki et al. (39) describieron que las orizacistatinas OC-I y OC-II reducían la replicación del virus del herpes simple (HSV-1), probablemente debido a una inhibición de proteasas víricas implicadas en el procesamiento de la cápside del mismo. Además, Gutierrez-Campos et al. (40) comprobaron que plantas de tabaco que expresan la OC-I presentaban cierto grado de resistencia frente a infecciones de TMV, y recientemente se ha demostrado un ligero efecto inhibitorio de varias fitocistatinas sobre las cisteín-proteasas de un potivirus (41). No obstante, es necesario que las fitocistatinas presenten mayor afinidad y especificidad por las proteasas víricas para ser buenos candidatos como moléculas antivirales.
En 1999 se describió por primera vez la actividad antifúngica de una cistatina de castaño como inhibidor del crecimiento de los hongos fitopatógenos Botrytis cinerea , Colletotrichum graminicola y Septoria nodorum y del saprofito Trichoderma viride (42), cuyos mRNAs se acumulan en hojas inoculadas con esporas de B. cinerea .
Posteriormente, se han demostrado las propiedades fungicidas de fitocistatinas de caña de azúcar, cebada, fresa y taro (3, 9, 43, 44). Sin embargo, aún hoy se desconoce el mecanismo de acción de esta actividad. Se ha comprobado mediante mutagénesis dirigida de la cistatina HvCPI-1 de cebada que los dominios de la proteína implicados en la inhibición enzimática no son los responsables de su actividad antifúngica (9). Además, la cistatina HvCPI-1 impide el crecimiento in vitro de B. cinerea, pero no se ha podido detectar la presencia de cisteín-proteasas ni en extractos proteicos del micelio ni del medio de cultivo del hongo (9). Recientemente, se han observado alteraciones en la permeabilidad de las membranas de B. cinerea y Fusarium oxysporum crecidos en presencia de HvCPI-1 (Martinez et al., datos sin publicar). Sin embargo, la cistatina de taro que presenta un claro efecto tóxico sobre el crecimiento del micelio del hongo fitopatógeno Sclerotium rolfsii es capaz de inhibir, a la vez, las cisteín-proteasas de extractos de dicho micelio (44).
Otro argumento para implicar a las fitocistatinas en el sistema de defensa de la planta es su inducción por herida y ácido jasmónico. El daño generado por una herida mecánica o por insectos fitófagos produce una acumulación de inhibidores de proteasas tanto a nivel local como de forma sistémica. Además, se sabe que el ácido jasmónico es una fitohormona implicada en la transducción de señales que median la respuesta de la planta a la herida y al estrés biótico. Se han identificado cistatinas que se inducen tanto por herida como por metil jasmonato, y se ha descrito que dichos tratamientos producen un aumento paralelo de la actividad inhibitoria frente a papaína (23, 27). En tomate, sin embargo, se han caracterizado y purificado cistatinas inducibles por ácido jasmónico, cuyos niveles de expresión no responden a herida (6).
Otros tratamientos abióticos como el estrés salino, la anaerobiosis, la sequía y la incubación a altas o bajas temperaturas, también inducen la expresión de fitocistatinas (17, 20, 23).
Uno de los mayores problemas de la agricultura moderna son los daños causados por especies fitopatógenas que producen pérdidas anuales de las cosechas estimadas en un 18% en el caso de los insectos fitófagos y casi un 20% debido a enfermedades bacterianas y fúngicas. El control de plagas de cultivos y productos almacenados se realiza de forma mayoritaria mediante la utilización de plaguicidas orgánicos de síntesis, cuyo uso mejora la producción pero provoca la aparición de graves problemas medioambientales. La necesidad de desarrollar métodos de control alternativos ecológicamente aceptables es cada vez más apremiante y una de las estrategias de lucha ha consistido en el aislamiento, transferencia y expresión de genes con capacidad insecticida y/o fungicida de origen natural, en especies vegetales de interés agronómico. La tecnología del DNA recombinante y la transgénesis vegetal han permitido que esta estrategia sea posible.
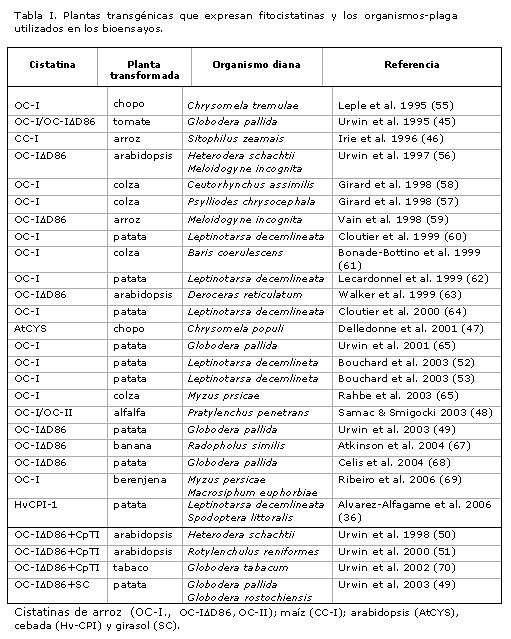
Muchos son los ejemplos publicados durante la última década en los que se han utilizado fitocistatinas como transgenes insecticidas y se ha demostrado que las plantas establemente transformadas con estos genes de origen vegetal presentan un aumento de la resistencia contra insectos, nematodos, virus y babosas (Tabla I).
La fitocistatina más utilizada como transgén de defensa ha sido la orizacistatina I de arroz, OC-I, y una variante de la misma, la OC-I D D 86 generada por mutagénesis dirigida, en la que se ha eliminado el residuo 86 de su secuencia de aminoácidos correspondiente a un ácido aspártico, confiriéndole mayor capacidad de inhibir cisteín-proteasas (45). OC-I y su variante OC-I D D86, se han expresado en al menos nueve especies vegetales distintas (patata, tomate, arroz, arabidopsis, chopo, banana, colza, alfalfa y berenjena; Tabla I) con las que se han realizado bioensayos con distintos organismos. Larvas de coleópteros, áfidos, nemátodos y babosas alimentadas con estas especies transgénicas han mostrado alteraciones en sus enzimas digestivos que se han traducido en una disminución del peso, y han afectado a su desarrollo, fecundidad y supervivencia, dificultando en muchos casos que se complete el ciclo vital de estos organismos plaga y por tanto, protegiendo las plantas de sus efectos nocivos.
Aparte de la mencionada cistatina OC-I de arroz, de las más de 80 fitocistatinas identificadas, solo las cistatinas OC-II de arroz, CC-I de maíz, AtCYS de arabidopsis, SC de girasol y HvCPI-1 de cebada se han expresado transgénicamente en plantas (36, 46-49). Los efectos producidos por las correspondientes proteínas expresadas en arroz, chopo, alfalfa y patata han demostrado su papel de defensa contra los coleópteros Sitophilus zeamais , Chrysomela populi y Leptinotarsa decemlineata y nemátodos del género Globodera .
En algunos casos la presencia del transgén insecticida o los niveles de expresión de la proteína que codifican, no son suficientes para alcanzar el grado de protección deseado. La estrategia denominada “gene pyramiding”, que combina la expresión de múltiples transgenes insecticidas en una misma especie vegetal y la expresión bajo el control de un promotor de dos genes insecticidas o fragmentos fusionados de los mismos han conseguido aumentar la protección y a la vez reducir los riesgos de adaptación y el desarrollo de resistencia de los insectos (49-51). Un ejemplo representativo de las ventajas que aporta esta estrategia se muestra en la resistencia aditiva frente al nematodo Globodera que presentan las patatas que co-expresan la orizacistatina OC-I D D86 y la cistatina CEWC (de clara de huevo de pollo) y la combinación de cistatinas SC de girasol con CEWC, en ambos casos comparada con las resistencias parciales de cada gen expresado independientemente (49).
Aunque como se ha descrito anteriormente, ensayos de inhibición in vitro frente a hongos fitopatógenos han demostrado que las fitocistatinas también poseen propiedades antifúngicas, todavía no se ha comprobado su efecto en plantas transgénicas.
Finalmente, es importante mencionar el incremento de publicaciones que se han centrado en analizar los posibles efectos de los transgenes insecticidas sobre organismos no plaga ecológicamente importantes para el medio, como son los enemigos naturales y los parasitoides. Este tipo de estudios ha incluido a las fitocistatinas y hasta la fecha ningún inhibidor de cisteín-proteasas de origen vegetal utilizado como transgén ha mostrado efectos negativos en las interacciones tritróficas analizadas (36, 52-54).
En conclusión, todos estos estudios refuerzan el papel de estas proteínas en la defensa vegetal, y confirman su potencial biotecnológico para la obtención de plantas resistentes a organismos fitopatógenos.
 |
Los autores agradecen al Ministerio de Educación y Ciencia la financiación aportada (proyecto BFU2005-00603/BMC) para llevar a cabo la investigación vinculada al tema desarrollado.
1. Rassam M, Laing WA . 2004. Purification and characterization of phytocystatins from kiwi cortex and seeds. Phytochemistry, 65: 19-30.
2. Lim CO, Lee SI, Chung WS et al. 1996. Characterization of a cDNA encoding a cysteine proteinase inhibitor from Chinese cabbage ( Brassica campestris L. ssp. pekinensis ) flower buds. Plant Molecular Biology, 30: 373-379.
3. Martinez M, Abraham Z, Gambardella M et al. 2005. The strawberry gene Cyf1 encodes a phytocystatin with antifungal properties. Journal of Experimental Botany, 56: 1821-1829.
4. Martinez M, Abraham Z, Carbonero P et al. 2005. Comparative phylogenetic analysis of cystatin gene families from arabidopsis, rice and barley. Molecular Genetics and Genomics, 273: 423-432.
5. Waldrom C, Weigrich LM, Merlo PAO et al. 1993. Characterization of a genomic sequence coding for potato multicystatin, an eight domain cysteine proteinase inhibitor. Plant Molecular Biology, 23: 801-812.
6. Wu J, Haard NF . 2000. Purification and characterization of a cystatin from leaves of methyl jasmonate treated tomato plants. Comparative Biochemistry and Physiology, 127: 209-220.
7. Nagata K, Kudo N, Abe K et al. 2000. Three-dimensional solution structure of oryzacystatin-I, a cysteine proteinase inhibitor of rice , Oryza sativa L. japonica. Biochemistry, 39: 181-201.
8. Belenghi B, Acconcia F, Trovato M et al. 2003. AtCYS1, a cystatin from Arabidopsis thaliana , suppresses hypersensitive cell death. European Journal of Biochemistry, 270: 2593-2604.
9. Martinez M, Lopez-Solanilla E, Rodriguez-Palenzuela P et al. 2003. Inhibition of plant-pathogenic fungi by the barley cystatin HvCPI (gene Icy ) is not associated with its cysteine-proteinase inhibitory properties. Molecular Plant-Microbe Interactions, 16: 876-883.
10. Stuff MT , Laber B, Bode W et al. 1990. The refined 2.4 A X-ray crystal structure of recombinant human stefin B in complex with the cysteine proteinase papain: a novel type of proteinase inhibitor interaction. The EMBO Journal, 9: 1939-1947.
11. Jenko S, Dolenc I, Dobersek A et al. 2003. Crystal structure of stefin A in complex with cathepsin H: N-terminal residues of inhibitors can adapt to the active sites of endo- and exopeptidases. Journal of Molecular Biology, 326: 875-885.
12. Abe M, Kondo H, Arai S. 1987. Purification and characterization of a rice cysteine proteinase inhibitor. Agricultural and Biological Chemistry, 51: 2763-2768.
13. Kuroda M, Kiyosaki T, Matsumoto I et al. 2001. Molecular cloning, characterization and expression of wheat cystatins, Bioscience Biotechnology and Biochemistry, 65 : 22-28.
14. Corre-Menguy F, Cejudo FJ, Mazubert C et al. 2002. Characterization of the expression of a wheat cystatin gene during caryopsis development. Plant Molecular Biology, 50: 687-698.
15. Abe M, Domoto C, Watanabe H et al. 1996. Structural organization of the gene encoding corn cystatin. Bioscience, Biotechnology and Biochemistry 60:1173-1175.
16. Kondo H, Abe K, Nishimura I et al. 1990. Two distinct cystatin species in rice seeds with different specificities against cysteine proteianses. Journal of Biological Chemistry, 265: 16832-15837.
17. Gaddour K, Vicente-Carbajosa J, Lara P et al. 2001. A constitutive cystatin-encoding gene from barley ( Icy ) responds differentially to abiotic stimuli. Plant Molecular Biology, 45: 599-608.
18. Li Z, Sommer A, Dingermann T et al. 1996. Molecular cloning and sequence analysis of a cDNA encoding a cysteine proteinase inhibitor form Sorghum bicolor seedlings. Molecular General Genetics, 251: 499-502.
19. Martinez M, Abraham Z, Carbonero P et al. 2004. Plant cystatins: a genomic approach in rice and barley. En: Challenges and opportunities for sustainable rice-based production systems (Ferrero A & Vidotto F eds). Edizioni Mercurio. Vercelli , Italy pag. 371-377.
20. Massonneau A, Confamine P, Wisniewski JP et al. 2005. Maize cystatins respond to developmental cues, cold stress and drought. Biochemica et Biophysica Acta, 1729: 186-199.
21. Reis EM, Margis R. 2001. Sugarcane phytocystatins: identification, classification and expression pattern. Genetics and Molecular Biology, 24: 291-296.
22. Pernas M, Sanchez-Monge R, Gomez L et al. 1998. A chestnut seed cystatin differentially effective against cysteine proteinase from closely related pests. Plant Molecular Biology, 38: 1235-1242.
23. Pernas M, Sanchez-Monge R, Salcedo G. 2000. Biotic and abiotic stress can induce cystatin expression in chestnut. FEBS Letters, 467: 206-210.
24. Martinez M, Rubio-Somoza I, Fuentes R et al. 2005. The barley cystatin gene ( Icy ) is regulated by DOF transcription factors in aleurone cells upon germination. Journal of Experimental Botany, 56: 547-556.
25. Ryan SN, Laing WA , McManus MT. 1998. A proteinase inhibitor purified from apple fruit. Phytochemistry, 49: 957-963.
26. Ojima A, Shiota H, Higashi K et al. 1997. An extracellular insoluble inhibitor de cysteine proteinases in cell cultures and seed of carrots. Plant Molecular Biology, 34: 99-109.
27. Grunden K, Strukelj B, Ravnikar M et al. 1997. Potato cysteine proteinase inhibitor gene family: molecular cloning, characterization of avocado fruit mRNAs and their expression during ripening and low-temperature storage. Plant Molecular Biology, 34: 317-323.
28. Madureira HC, Da Cunha M, Jacinto T. 2006. Immunolocalization of a defense-related 87 kDa cystatin in leaf blade of tomato plants. Environmental and Experimental Botany, 55: 201-208.
29. Steller H. 1995. Mechanisms and genes of cellular suicide. Science, 267: 1445-1449.
30. Arai S, Matsumoto I, Emori Y et al. 2002. Plant seed cystatins and their target enzymes of endogenous and exogenous origin. Journal of Agricultural Food and Biochemistry, 50: 6612-6617.
31. Martinez M, Rubio-Somoza I, Carbonero P et al. 2003. A cathepsin B-like cysteine protease gene from Hordeum vulgare (gene CatB ) induced by GA in aleurone cells is under circadian control in leaves. Journal of Experimental Botany, 54: 951-959.
32. Song I, Taylor M, Baker K et al. 1995. Inhibition of cysteine proteinases by Carica papaya cystatin produced in Escherichia coli . Gene, 162: 221-224.
33. Solomon M, Belenghi B, Delledonne M et al. 1999. The involvement of cysteine proteases and protease inhibitor genes in the regulation of programmed cell death in plants. The Plan Cell, 11: 431-443.
34. Gholizadeh A, Santa IM, Kohnchrouz BB et al. 2005. Cystatin may confer viral resistance in plants by inhibition of a virus-induced cell death phenomenon in which cysteine poteinases are active: cloning and molecular characterization of a cDNA encoding cysteine proteinase inhibitor (celostatin) from Celosia cristat a (crested cock´s comb). Biotechnology and Applied Biochemistry, 42: 197-204.
35. Koiwa H, Shade RE, Zhu-Salzman K et al. 2000. A plant cystatin (soyacystatin) targets cathepsin L-like digestive cysteine proteinasee (DvCALs) in the larval midgut of western corn rootworm ( Diabrotica virgifera virgifera ). FEBS Letters, 471: 67-70.
36. Alvarez-Alfageme F, Martinez M, Pascual-Ruiz S et al. 2006. Effects of potato plants expressing a barley cystatin on the predatory bug Podisus maculiventris via herbivorous prey feeding on the plant. Transgenic Research (en prensa).
37. Aguiar JM, Franco OL, Rigden DJ et al. 2006. Molecular modelling and inhibitory activity of cowpea cystatin against bean bruchid pests. Proteins: structure, function, and bioinformatics (en prensa).
38. Pernas M, Sanchez-Ramos I, Sanchez-Monge R et al. 2000b. Der p1 and Der f1, the highly related and major allergens from house mites, are differentially affected by a plant cystatin. Clinical Experimental Allergy, 30: 792-978.
39. Aoki H, Akaike T, Abe K et al. 1995. Antiviral effect of oryzacystatin, a proteinase inhibitor in rice, against herpes simplex virus type 1 in vitro and in vivo. Antimicrobial Agents and Chemotherapy, 39: 846-849.
40. Gutierrez-Campos R, Torres-Acosta JA, Saucedo-Arias LJ et al. 1999. The use of cysteine proteinase inhibitors to engineer resistance against potyviruses in transgenic tobacco plants. Nature Biotechnology, 17: 1223-1226.
41. Wen R, Zhang SC, Michaud D et al. 2004. Inhibitory effects of cystatins on proteolytic activities of the Plum pox potyvirus cysteine proteinases. Virus Research, 105: 175-182.
42. Pernas M, Lopez-Solanilla E, Sanchez-Monge R et al. 1999. Antifungal activity of a plant cystatin. Molecular Plant-Microbe Interactions, 12: 624-627.
43. Soares-Costa A, Beltramini LM, Thiemann OH et al. 2002. A sugarcane cystatin: recombinant expression, purification and antifungal activity. Biochemical and Biophysical Research Communications, 206: 1194-1199.
44. Yang AH, Yeh KW. 2005. Molecular cloning, recombinant gene expression, and antifungal activity of cystatin from taro ( Colocasia esculenta cv. Kaosiung no.1). Planta, 221: 493-501.
45. Urwin PE, Atkinson HJ, Waller DA et al. 1995. Engineered oryzacystatin-I expressed in transgenic hairy roots confers resistance to Globodera pallida . The Plant Journal, 8: 121-131.
46. Irie K, Hosoyama H, Takecuchi T et al. 1996. Transgenic rice established to express corn cystatin exhibits strong inhibitory activity against insect gut proteinases. Plant Molecular Biology, 30: 149-157.
47. Delledonne M, Allegro G, Belenghi B et al. 2001. Transformation of white poplar ( Populus alba L.) with a novel Arabidopsis thaliana cysteine proteinase inhibitor and analysis of insect pest resistance. Molecular Breeding, 7: 35-42.
48. Samac DA, Smigocki AC. 2003. Expression of oryzacystatin I and II in alfalfa increases resistance to the root-lesion nematode. Nematology, 93: 799-804.
49. Urwin PE, Green J, Atkinson HJ. 2003. Expression of a plant cystatin confers partial resistance to Globodera , full resistance is achieved by pyramiding a cystatin with natural resistance. Molecular Breeding, 12: 263-269.
50. Urwin PE, McPherson MJ, Atkinson HJ. 1998. Enhanced transgenic plant resistance to nematodes by dual proteinase inhibitor constructs. Planta, 204: 472-479.
51. Urwin PE, Levesley A, McPherson MJ et al. 2000. Transgenic resistance to the nematode Rotylenchulus reniformis conferred by Arabidopsis thaliana plants. Molecular Breeding, 6: 257-264.
52. Bouchard E, Cloutier C, Michaud D. 2003. Oryzacystatin I expressed in transgenic potato induces digestive compensation in an insect natural predator via its herviborous prey feeding on the plant. Molecular Ecology, 12: 2439-2446.
53. Bouchard E, Michaud D, Cloutier C. 2003. Molecular interaction between an insect predator an its hervibore prey on transgenic potato expressing a cysteine proteinase inhibitor from rice. Molecular Ecology, 12: 2429-2437.
54. Cowgill SE, Danks C, Atkinson HJ. 2004. Multitrophic interactions involving genetically modified potatoes, nontarget aphids, natural enemies and hyperparasitoids. Molecular Ecology, 13: 639-647.
55. Leple JC, Bonade-Bottino M, Augustin S et al. 1995. Toxicity to Chrysomela tremulae (Coleoptera: Chrysomelidae) of transgenic poplars expressing a cysteine proteinase inhibitor. Molecular Breeding, 1: 319-328.
56. Urwin PE, Lilley CJ, McPherson MJ et al. 1997. Resistance to both cyst and root-knot nematode conferred by transgenic Arabidopsis expressing a modified plant cystatin. The Plant Journal, 12: 455-461.
57. Girad C, Bonade-Bottino M, Pham-Delegue M-H et al. 1998. Two starins of cabbage seed weevil (Coleoptera: Curculionidae) exhibit differential susceptibility to a transgenic oilseed rape expressing oryzacystatin I. Journal of Insect Physiology, 44: 569-577.
58. Girad C, Le Metayer M, Zaccomer B et al. 1998. Growth stimulation of beetle larvae reared on a transgenic oilseed rape expressing a cysteine proteinase inhibitor. Journal of Insect Physiology, 44: 263-270.
59. Vain P, Worland B, Clarke MC et al. 1998. Expression of an engineered cysteine proteinase inhibitor (oryzacystatin-I D D86) for nematode resistance in transgenic rice plants. Theoretical and Applied Genetics, 96: 266-271.
60. Cloutier C, Fournier M, Jean C et al. 1999. Growth compensation and faster development of Colorado potato beetle (Coleoptera : Chrymelidae) feeding on potato foliage expressing oryzacystatin I. Archives of Insect Biochemistry and Physiology, 40: 69-79.
61. Bonade-Bottino M, Lerin J, Zaccomer B et al. 1999. Physiological adaptation explains the insensitivity of Baris coerulescens to transgenic oilseed rape expressing oryzacystatin I. Insect Biochemistry and Molecular Biology, 29: 131-138.
62. Lecardonnel A, Chauvin L, Jouanin L et al. 1999. Effects of rice cystatin I expression in transgenic potato on Colorado potato beetle larvae. Plant Science, 140: 71-79.
63. Walker AJ, Urwin PE , Atkinson HJ et al. 1999. Transgenic Arabidopsis leaf tissue expressing a modified oryzacystatin shows resistance to the field slug Deroceras reticulatum (Muller). Transgenic Research, 8: 95-103.
64. Cloutier C, Jean C, Fournier M et al. 2000. Adult Colorado potato beetles, Leptinotarsa decemlineata compensate for nutritional stress on oryzacystatin I-transgenic potato plants by hypertrophic behaviour and over-production of insensitive protease. Archives of Insect Biochemistry and Physiology, 44: 69-81.
65. Urwin PE, Troth KM, Zubko EI et al. 2001. Effective transgenic resistance to Globodera pallida in potato field trials. Molecular Breeding, 8: 95-101.
66. Rahbe Y, Derason C, Bonade-Bottino M et al. 2003. Effects of the cysteine protease inhibitor oryzacystatin (OC-I) on different aphids and reduced performance of Myzus persicae on OC-I expressing transgenic oilseed rape. Plant Science, 164: 441-450.
67. Atkinson HJ, Grimwood S, Johnston K et al. 2004. Prototype demonstration of transgenic resistance to the nematode Rodopholus similis conferred on banana by a cystatin. Transgenic Research, 13: 135-142.
68. Celis C, Scurrah M, Cowgill S et al. 2004. Environmental biosafety transgenic potato in a centre of diversity for this crop. Nature, 432: 222-225.
69. Ribeiro APO, Pereira EJC, Galvan TL et al. 2006. Effect of eggplant transformed with oryzacystatin gene on Myzus persicae and Macrosiphum euphorbiae . Journal of Applied Entomology, 130: 84-90.
70. Urwin PE, Zubko EI, Atkinson HJ. 2002. The biotechnological application and limitation of IRES to deliver multiple defense genes to plant pathogens. Physiological and Molecular Plant Pathology, 61: 103-108.