Al igual que otros organismos, las plantas se encuentran sometidas al ataque de numerosos y diferentes microorganismos patógenos, que incluyen nematodos, virus, bacterias y hongos (figura 1). Tanto las partes aéreas como las subterráneas están en continuo contacto con potenciales agentes patógenos. Por ello, no nos sorprenden las pérdidas causadas por las plagas en cosechas y cultivos. Y más de una vez habremos presenciado como algunas de las plantas que tenemos en casa sufren enfermedades y ataques de insectos y larvas. Cada especie vegetal está afectada aproximadamente por más de 100 tipos distintos de hongos, bacterias, virus y nematodos. En el medio natural cada planta suele ser atacada por varios cientos o miles de individuos de un mismo tipo de patógeno. A pesar de que estas plantas resultan en menor o mayor grado dañadas, muchas de ellas sobreviven a estos ataques y algunas de ellas continúan desarrollándose y multiplicándose.
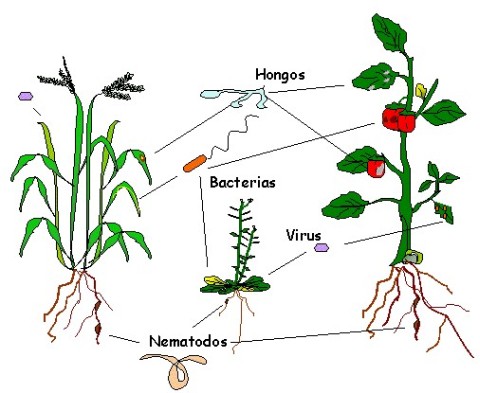
Figura 1. La mayoría de los patógenos ataca sólo una parte específica de la planta y produce una serie de síntomas característicos, tales como necrosis, manchas, secado de hojas, alteraciones en las raíces. Algunas especies de patógenos, en su mayoría hongos y nematodos, penetran directamente las capas superficiales por presión mecánica o mediante ataque enzimático. Otras especies pasan a través de aperturas naturales (como los estómas y lenticelas). Un tercer grupo invade sólo el tejido que previamente ha sido dañado. Mientras que otros patógenos son capaces de alcanzar el interior de la planta a través de vectores, es el caso de muchos virus que son introducidos directamente en el floema de muchas plantas por la acción de insectos y otras especies chupadoras tales como pulgones. Para evitar el ataque de estos patógenos la planta debe ser capaz de detectarlos tan rápidamente como sea posible, con objeto de evitar su rápida proliferación. El tiempo que tarde la planta en detectarlos, va a determinar, en gran medida, el éxito frente al patógeno.
Exceptuando la posibilidad de desplazamiento, que les permitiría buscar ambientes más favorables. Las plantas difieren de los animales en dos aspectos que determinan profundamente las estrategias a seguir para defenderse de los patógenos. En primer lugar, las plantas carecen de un sistema inmunológico, de forma que no existen células especializadas para defender el organismo, cada célula individual debe tener la capacidad de responder al ataque del patógeno. En segundo lugar, las plantas pueden perder porciones considerables de su estructura sin que ello traiga consigo problemas de supervivencia, pudiendo inducir la muerte de órganos invadidos por el patógeno para evitar la propagación de este al resto de la planta, y este mecanismo de defensa conocido como respuesta hipersensible (HR) es el más utilizado por las plantas para defenderse frente a los organismos patógenos. Este tipo de defensa puede a su vez activar otros mecanismos más duraderos y ventajosos para la planta, como es la denominada resistencia sistémica adquirida (SAR), que consiste en la generación de resistencias frente a futuros patógenos de células de la planta que no han estado en contacto con este.
El proceso de interacción entre huésped y patógeno es un proceso dinámico en el que ambos intervienen (1). Cada patógeno ha evolucionado de una forma distinta en cuanto al modo en que invade a su planta huésped, y la existencia de distintas barreras de defensa en la planta junto con las condiciones ambientales presentes en un determinado momento, condicionan el éxito del patógeno en cuanto a su colonización y proliferación en el huésped.
• En general, las plantas se defienden contra estos organismos mediante una combinación de características estructurales y bioquímicas, que se conoce como defensa pasiva, que varía con el estado de desarrollo de la planta, tipo de órgano y tejido afectado.
• A veces, la especie vegetal afectada por el patógeno es incapaz de mantener la estrategia de crecimiento y multiplicación de un patógeno en particular y por lo tanto el patógeno no la reconoce como huésped.
• Las condiciones nutricionales y medioambientales en que se encuentran las plantas pueden fluctuar y cambiar de forma que afecten la capacidad de invasión del patógeno.
Además de las barreras de defensa presentes antes de la infección del patógeno en la planta, la defensa pasiva, el reconocimiento del patógeno induce la activación de nuevas respuestas de defensa que permiten restringir el sitio de infección, y el refuerzo de otras barreras previas ya existentes, lo que constituye la defensa activa.
La primera línea de defensa de las plantas frente al patógeno la constituye su propia superficie, que el patógeno debe penetrar para producir la infección. Las defensas estructurales presentes en la planta antes de que se produzca el reconocimiento del patógeno están constituidas por la cantidad y tipo de ceras que cubren las células epidérmicas, la estructura de la pared celular de éstas células, el tamaño, localización y forma de los estomas y lenticelas, y la presencia de estructuras especiales en la epidermis que dificultan la penetración de patógeno tales como pelos y espinas.
Las ceras presentes en la superficie de hojas y frutos actúan como repelentes del agua previniendo la formación de una película de humedad en al que los patógenos podrían depositarse, germinar (hongos) y multiplicarse (bacterias). Una zona muy tupida de pelos en la superficie de la planta puede ejercer este mismo efecto de repelente de agua y por lo tanto reducir la posibilidad de colonización de la superficie por parte del patógeno. Una cutícula gruesa podría incrementar la resistencia a la infección en aquellos casos en los que el patógeno sólo es capaz de producir infección cuando penetra en el interior del huésped. El grosor y composición de la pared celular son factores importantes en la generación de resistencias a la entrada de patógenos. La presencia de paredes gruesas dificulta la penetración de hongos, si bien estos pueden alcanzar el interior a través de heridas presentes en la superficie de la planta.
Las defensas químicas presentes en la planta antes de que se produzca el reconocimiento del patógeno resultan mucho más efectivas que las defensas estructurales. Las plantas exudan una amplia variedad de sustancias a través de su superficie, especialmente en el caso de las raíces. Algunos de los compuestos secretados son producidos exclusivamente por determinadas especies de plantas y parecen tener una función de inhibidores frente a determinado tipo de patógenos (2). Determinados exudados producidos en la superficie de las hojas son tóxicos para hongos, es el caso de los producidos por las plantas de tomate y azúcar de caña, que se produce en cantidades suficientes como para inhibir la germinación de las esporas de los hongos Botrytis y Cercospora, respectivamente. Así mismo, en el caso de determinadas variedades de cebolla resistentes al hongo Colletotricum circinans se ha comprobado la secreción de compuesto fenólicos no producidos por las variedades susceptibles de cebolla. La presencia de fenoles y taninos, y de algunos compuestos derivados de ácidos grasos como algunos dienos, presentes a elevadas concentraciones en frutos jóvenes, hojas o en semillas, han sido propuestos como los responsables de las resistencias de ciertos tejidos más jóvenes a organismos fitopatógenos. Muchos de estos compuestos actúan como inhibidores de muchas enzimas hidrolíticas fúngicas. Otros compuestos con actividad inhibitoria son las saponinas (compuestos esteroides y triterpenoides glucosilados) (3,4), la tomatina en tomate y la avenacina en avena, presentan actividad lítica sobre membranas. Además de estos compuestos, se han descrito determinadas proteínas vegetales que actúan como inhibidoras de proteinasas del patógeno o de enzimas hidrolíticas que degradan la pared celular de la planta, que inactivan ribosomas no reconocidos como propios de la planta, o que actúan incrementando al permeabilidad de la membrana fúngica. De forma similar, las lectinas, proteínas que unen de forma específica cierto tipo de azúcares, y que se presentan en alta concentración en muchos tipos de semillas, producen lisis e inhibición del crecimiento de muchos tipos de hongos. Así mismo, las glucanasas y quitinasas presentes en las secreciones y en la superficie de las células vegetales actúan degradando la pared celular del patógeno, contribuyendo a generar resistencias frente a la infección (5).
Sólo una pequeña proporción de las interacciones entre las plantas y los patógenos acaban en la infección y colonización de la planta huésped. Muchas plantas sólo hacen uso de una defensa activa, inducida una vez se ha detectado la presencia del patógeno, en vez de poseer un buen arsenal de elementos de la defensa pasiva. La explicación de este mecanismo de defensa se basa en la optimización de los recursos de la planta, en vez de invertir en la creación de un sistema de defensa preestablecido, prefiere invertir dichos recursos en su desarrollo, crecimiento y multiplicación. La planta activa un sistema de defensa sólo cuando es realmente necesario (6).
Las plantas tienen la capacidad de reconocer la presencia en el medio de microorganismos potencialmente patogénicos y de activar una respuesta de defensa eficiente para su eliminación (7,8) (Figura 2). La activación de una defensa eficaz por parte de la planta se basa en la rapidez con la que es capaz de detectar la presencia del patógeno.
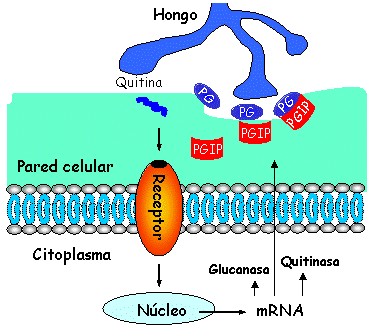
Figura 2. El reconocimiento de las moléculas elicitoras por parte de los receptores de la planta, trae consigo la activación de una cascada de señales dirigidas a la puesta en marcha de los mecanismos de defensa frente al establecimiento y multiplicación del patógeno. Las paredes celulares actúan de barrera física entre el patógeno y la célula vegetal. Además, las paredes celulares actúan como almacén de numerosos metabolitos secundarios y proteínas antimicrobianas de la planta, que inhiben el crecimiento de muchos patógenos. En la figura, el hongo produce una serie de enzimas hidrolíticas, poligalacturonasas y peptato liasas, que degradan los polisacáridos de la pared de la planta, esta degradación trae consigo la liberación de sustancias que afectan el desarrollo del hongo. Así por ejemplo las quitinasas de la planta actúan degradando la quitina del hongo, que es a su vez reconocida por la planta como una molécula elicitora, indicadora de la presencia del patógeno. La unión del elicitor a su receptor activa una respuesta de defensa en la célula vegetal.
Las plantas emplean una elevada cantidad de señales originadas por los microorganismos y el ambiente, que les permiten reconocer al patógeno y activar sus mecanismos de defensa (9). Estos compuestos se conocen con el nombre de elicitores. Hay dos clases de elicitores: los elicitores no específicos, inducen respuestas de defensa en un amplio rango de especies vegetales. Entre este tipo de elicitores se encuentran los fragmentos de paredes celulares, enzimas hidrolíticos producidos por la planta o por el patógeno durante el proceso de infección, ciertos péptidos, glicoproteínas y ácidos grasos polinsaturados (10). Este tipo de elicitores inducen respuestas de defensa en un gran número de especies vegetales. La segunda clase de elicitores la constituyen los elicitores específicos. Estos inducen la reacción de defensa contra un patógeno muy específico, y este tipo de elicitores es producido únicamente por el patógeno. Los genes que codifican para estos elicitores se conocen con el nombre de genes de avirulecia (Avr), encontrándonos que dentro de una determinada especie de patógenos, sólo unos pocos son capaces de producir este tipo de elicitores (11). La resistencia de una planta frente a un patógeno que posea este tipo de gen de avirulencia, sólo es posible si la planta posee un gen (genes de resistencia, genes R), cuyo producto es capaz de reconocer el producto del gen Avr, y por lo tanto de activar la respuesta de defensa (figura 3). Las bases genéticas en la planta, que determinan el proceso de resistencia frente a elicitores específicos, están basadas en la presencia de los llamados "genes de resistencia" (R), presentes en la planta, y de los correspondientes "genes de avirulencia" (Avr), presentes en el patógeno (12). De esta forma, la respuesta de defensa existe como una consecuencia de la interacción específica entre el producto de un gen R y el producto de un gen Avr. La existencia de este tipo de relación fue propuesta por Flor cuando enunció su famosa hipótesis del reconocimiento "gen a gen" (13).
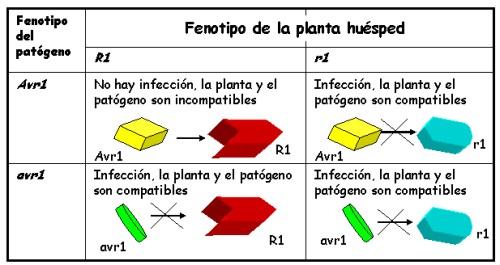
Figura 3. Los elicitores específicos permiten que la planta sea capaz de defenderse y resistir a determinado tipo de patógenos. La capacidad de resistir al patógeno está condicionada por la presencia en el patógeno de los llamados genes de avirulencia, y en la planta del correspondiente gen de resistencia. Los genes de avirulencia determinan los huéspedes que el patógeno infecta. La existencia de este tipo de interacción sugiere una competición evolutiva en la que el patógeno produce genes de virulencia, y la planta como respuesta desarrolla genes R que reconocen los productos de los genes de virulencia, que una vez reconocidos pasan a ser genes de avirulencia. La no existencia en la planta del correspondiente gen R, imposibilita la detección del patógeno por parte de la planta y su capacidad de resistir al ataque del mismo. De forma que durante el proceso evolutivo las plantas desarrollan sistemas de detección del patógeno mientras que estos desarrollan sistemas que les permitan evadir los sitemas de detección de la planta.
Aunque distintos, ambos tipos de elicitores son reconocidos por moléculas denominadas receptores presentes en la célula vegetal. El reconocimiento de estos elicitores activa toda una cascada de reacciones cuyo fin último es activar los mecanismos de defensa de la planta frente al patógeno, y por lo tanto se dice que la planta es resistente (14). Este proceso está temporal y espacialmente controlado para asegurar que sólo un número necesario de células serán activadas, y por lo tanto su metabolismo primario redirigido hacia la activación de la respuesta de defensa (15). Estas respuestas de defensa resultan en la creación de un medio exterior inapropiado para el mantenimiento y la proliferación del patógeno, y al mismo tiempo estas células serán capaces de desintoxicar y detener la acción de toxinas y otros compuestos generados por el patógeno. Esta activación de las respuestas de defensa tiene lugar dentro de las primeras 24 horas tras la infección del patógeno e invariablemente supone bien directa o indirectamente la muerte final de las células vegetales alrededor del sitio inicial de entrada del patógeno. La finalidad de este proceso de muerte celular es la restricción y confinamiento del patógeno en esa zona de la planta eliminando la disponibilidad de nutrientes para el patógeno (16).
Los estudios de interacción entre plantas y patógenos, han avanzado considerablemente durante los últimos años. Es un campo relativamente reciente en el que el empleo de la planta modelo Arabidopsis thaliana ha permitido un avance rápido y continuado, gracias al aislamiento e identificación de multitud de mutantes de esta planta que muestran alteraciones en su respuesta a la presencia de patógenos (17,18,19). Básicamente se han establecido dos estrategias de reconocimiento por parte de la planta. Por una parte cada célula de la planta es capaz de percibir la presencia de moléculas extrañas y ajenas a ella. Y por otro lado cada célula es capaz de percibir cualquier cambio en alguna de sus propias moléculas. La gran mayoría de los microorganismos patógenos son no invasivos permaneciendo en el exterior de las superficies, donde establecen colonias con un elevado número de bacterias sin que causen ningún tipo de infección. Bajo condiciones ambientales adecuadas, estas bacterias alcanzan las partes internas de la planta a través de entradas naturales, como los estomas o a través de zonas dañadas. Alcanzando de esta forma los espacios intercelulares de la planta, en los que pretende sobrevivir y multiplicarse, pero encontrándose en contacto con los centinelas de las células vegetales.
Unos de los retos más importantes para la planta, es el de ser capaz de discriminar entre un elevado número de potenciales patógenos frente a aquellos que no lo son y frente a sí misma, mediante el uso de un número finito de receptores capaces de reconocer moléculas foráneas pertenecientes al patógeno. Para ello durante el proceso evolutivo, las plantas han desarrollo unos receptores que reconocen motivos altamente conservados sólo presentes en los patógenos (20) (figura 4). Estos motivos, denominados PAMPs (patrones moleculares asociados al patógeno), forman parte de moléculas que son imprescindibles para la supervivencia del patógeno, por lo que no están sujetos a rápidas tasas de cambio por mutaciones, que les permitirían escapar al reconocimiento por parte del receptor vegetal. Además, estas moléculas son compartidas por un elevado número de patógenos, lo que permite a la planta restringir el número de receptores necesarios para el reconocimiento de los mismos.
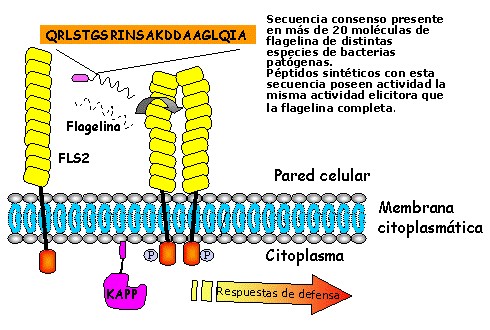
Figura 4. Modelo de reconocimiento y señalización desencadenado por el elicitor flagelina en Arabidopsis. Las bacterias se desplazan por la superficie de los tejidos de la planta haciendo uso del flagelo bacteriano. La presencia de heridas en la planta o de aperturas naturales, como son los estomas, permite el acceso de las bacterias al interior de los tejidos. En el espacio intercelular, las bacterias pierden moléculas de flagelina como consecuencia de roturas del flagelo y/o durante su construcción. La presencia de receptores FLS2 en la superficie de las células vestales permite la detección de la flagelina como indicador de la presencia de bacterias patógenas en el medio, lo que lleva a un desencadenamiento de las respuestas de defensa de la planta.
Durante los últimos años se han aislado y caracterizado numerosos elicitores inespecíficos en virus, hongos y bacterias. Estos elicitores, actúan como señales de la presencia del patógeno a muy bajas concentraciones, y algunos de ellos se han considerado dentro de la categoría de PAMPs (21). La percepción selectiva de estos por parte de la planta implica la existencia de receptores específicos. Al menos, para los elicitores de tipo hidrofílico se espera que estos receptores se encuentren situados a nivel de la membrana plasmática, y de hecho, se han identificado posibles sitios de unión de algunos de estos elicitores asociados a membranas vegetales (22,23). Este modelo de percepción en plantas, es paralelo a la inmunidad innata de animales (24), que así mismo está relacionada con el reconocimiento de PAMPs, que median las respuestas pro e inflamatorias.
El sistema de percepción de los PAMPs en plantas, está ejemplificado en el sistema de reconocimiento del elicitor bacteriano flagelina (25,26), también reconocida por animales e insectos como señal de la presencia de bacterias patógenas (27,28). La proteína flagelina es el componente principal del flagelo bacteriano, órgano locomotor de la bacteria, constituido por varios miles de moléculas de esta proteína. La construcción del flagelo bacteriano tiene lugar mediante el ensamblaje de moléculas de flagelina que van siendo secretadas al medio, de forma que si bien la mayoría de las moléculas de flagelina están formando parte del flagelo, la flagelina se acumula en el exterior como resultado de las roturas de los flagelos y durante la construcción de los mismos. Además de conferir movilidad, el flagelo permite a las bacterias responder a las condiciones favorables o desfavorables de su medio ambiente. La flagelina de las bacterias patógenas presenta actividad elicitora en distintas especies vegetales, siendo un elicitor inespecífico (29). La actividad elicitora de la molécula está localizada en el dominio amino terminal de la proteína, y más concretamente a una región de 25 aminoácidos que se encuentra altamente conservada entre las moléculas de flagelina de distintas especies de bacterias patógenas, pero que sin embargo difiere en el caso de las flagelinas de los flagelos de bacterias no patógenas, como es el caso de la bacteria Rhizobium que establece simbiosis con distintas especies de plantas (figura 4). Un péptido sintético, flg22, diseñado con la secuencia de aminoácidos de esta región conservada tiene la misma actividad elicitora que la molécula flagelina, demostrando con ello que es esta región la que específicamente reconocen las células vestales para detectar la presencia de bacterias patógenas. Este péptido sintético fue empleado para aislar e identificar el receptor responsable de su percepción en las células vegetales. Para proceder a tal identificación se hizo uso de la planta modelo Arabidopsis, se utilizó una población de mutantes con el objetivo de localizar plantas mutagenizadas que no reconocieran la péptido flg22, y que por lo tanto en presencia de este no respondiesen activando sus mecanismos de defensa. Como resultado de esta búsqueda se encontró la proteína FLS2. La proteína receptora FLS2, es una proteína transmembrana, que posee un largo dominio extracelular constituido por una serie de repeticiones ricas en el aminoácido leucina conocido con el nombre de LRR (repeticiones ricas en leucina), que participa en el reconocimiento y unión de la flagelina de bacterias patógenas; seguido de un dominio transmembrana que permite al receptor mantenerse anclado en la membrana de la célula vegetal; y un dominio intercelular quinasa, que es el responsable de transmitir la información al interior de la célula cuando se produce el reconocimiento de la flagelina. Esta serie de dominios estructurales presente en FLS2 también se presentan en las proteínas receptoras de animales e insectos que reconocen la flagelina de bacterias patógenas. La unión de la flagelina a FLS2 permite la activación de las respuestas de defensa en la planta frente a la bacteria patógena a través de una cascada de señalizaciones (30,31). Dos tipos de respuestas van a desencadenarse; una respuesta rápida que consiste en la alcalinización del medio extracelular, la generación de especies reactivas de oxígeno, la producción de la hormona etileno, que a su vez actúa de activador de otras respuestas de defensa, y deposición de calosa; la segunda respuesta implica la activación de la transcripción de genes que cuyos productos están implicados en la respuesta de defensa de la planta, bien de forma directa como es el caso de muchas proteínas PR, péptidos antimicrobianos, y defensinas (32) o bien participando en la amplificación de la señal de alarma en la planta.
Esta hipótesis se ha desarrollado a partir de los estudios de los mecanismos de reconocimiento de las proteínas Avr producidas por el patógeno por parte de los genes R de la planta. Hasta la fecha se han identificado docenas de genes R que confieren resistencias frente a patógenos muy diferentes (33). Estos genes codifican para proteínas que se pueden agrupar en varias superfamilias, basándose en la presencia de determinados dominios estructurales. La versión más sencilla expuesta en el caso anterior, es la de predecir una interacción directa entre la proteína Avr y la proteína R. Sin embargo, los numerosos experimentos llevados a cabo intentando demostrar tal interacción no han demostrado la existencia de dicha interacción, lo que ha llevado a la formulación de la “hipótesis del guardian” (34). Este modelo predice que las proteínas R activan una resistencia en las plantas cuando interaccionan con otra proteína de la planta “la protegida”, que es la molécula diana de las proteínas Avr del patógeno. Las proteínas R en el interior de las células se encargarían de vigilar activamente y en todo momento todos aquellos procesos celulares que son claves y son la diana para el patógeno. Puesto que dianas de las proteínas Avr del patógeno son probablemente bastante limitadas, este modelo explica como una planta puede detectar miles de patógenos con sólo unos pocos centenares de genes R. Esta hipótesis está avalada por trabajos publicados durante estos últimos tres años, entre ellas sirva de ejemplo el caso de la interacción del gen de resistencia Pto de tomate con la proteína AvrPto y AvrPtoB producida por la bacteria fitopatógena Pseudomonas syringae patovar tomato. El gen de resistencia que codifica a la proteína quinasa Pto fue el primer gen R que se clonó (35) , siendo uno de los genes mejor caracterizados y estudiados hasta la fecha (36). Las proteínas de avirulencia AvrPto y AvrPtoB, son sintetizadas en el patógeno e inyectadas por este en el interior de la célula vegetal mediante un complejo proteico que se conoce con el nombre de sistema de secreción tipo III, presente también en bacterias que infectan células animales, y cuya estructura es muy semejante a la del flagelo bacteriano (figura 5). Pto se localiza en el citoplasma de la célula vegetal próximo a la membrana plasmática, y por tanto próximo al lugar de entrada de las proteínas Avr (37). En un principio Pto se encuentra inactivo, es decir en ausencia del patógeno, Pto no está activando las señales que permiten a la planta defenderse del patógeno, la planta mantiene Pto inactivo mediante la unión en uno de los dominios de Pto de un péptido, que va a ser desplazado por AvrPto cuando sea inyectado en el interior de la célula vegetal. La unión de AvrPto trae consigo la activación de la actividad quinasa de Pto que se fosforila sufriendo por ello un cambio conformacional que es detectado por el guardian de Pto, la proteína de resistencia Prf presente también en el citoplasma de la célula vegetal (38,39). Es Prf la encargada de transmitir, junto a otras proteínas que se le unen la señal de alarma en la célula vegetal que activará los correspondientes mecanismos de defensa para resistir al patógeno.
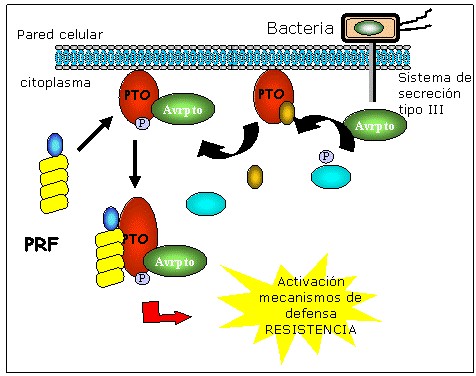
Figura 5. Modelo de activación de Pto por AvrPto en tomate. La proteína Pto está controlada por un péptido inhibitorio de su actividad, que actúa reprimiendo su actividad quinasa. Una vez que el elicitor Avrpto ha sido inyectado en el interior de la célula vegetal, AvrPto interacciona con Pto desplazando al péptido regulador del sitio catalítico y desreprimiendo la actividad quinasa. La proteína Pto activa cataliza su autofosforilación que genera un cambio conformacional de Pto, que es la señal de activación de la respuesta de defensa de la planta.
Las plantas han desarrollado y siguen desarrollando unos mecanismos de defensa tanto de tipo general, como altamente especializados, cuya finalidad es la de prevenir las enfermedades que podrían causarles todos aquellos potenciales patógenos con los que interaccionan constantemente. Hasta fechas muy recientes no se han comenzado a conocer e identificar los componentes directamente implicados en estos procesos de interacción planta-patógeno. En este artículo se han comentado principalmente los mecanismos de defensa frente a bacterias. Sin embargo, como ya indicamos al principio, las plantas también poseen mecanismos de defensa frente a otros microorganismos tales como hongos, virus y nemátodos. A pesar de las diferencias entre estos microorganismos y sus formas de vida, las plantas han desarrollado estrategias muy similares de detección y defensa. Curiosamente, también se han encontrado paralelismos entre los mecanismos empleados por muchos microorganismos patógenos de plantas y animales, y los últimos estudios moleculares están poniendo de manifiesto la existencia de similitudes en cuanto a las estrategias de defensa empleadas. Así pues, este campo de estudio podríamos considerarlo prácticamente como en sus inicios, y los estudios futuros se dirigen a la extrapolación de los resultados obtenidos con plantas modelo a plantas de interés agronómico, con el objeto de aplicar estos conocimientos al proceso de control d enfermedades y plagas.
1. Kniskern J, Rausher M. 2001. Two modes of host-enemy coevolution. Population Ecology, Vol 43:3-14.
2. Van Etten HD, Mansfield JW, Bailey JA, Farmer EE. 1994. Two classes of plant antibiotics: phytoalexins versus phytoanticipins. Plant Cell, Vol 6: 1191-1192.
3. Osbourn AE. 1996. Preformed antimicrobial compounds and plant defense against fungal attack. Plant Cell, Vol 8: 1821-1831.
4. Morrissey JP, Osbourn AE. 1999. Fungal resistance to plant antibiotics as a mechanism of pathogenesis. Microbiology Molecular Biology Reviews. vol 63:708-724.
5. Valueva TA, Mosolov VV. 2004. Role of Inhibitors of Proteolytic Enzymes in Plant Defense against Phytopathogenic Microorganisms. Biochemistry, Vol 69:1305-1309.
6. Fornoni J, Núñez-Farfán J, Valverde P-L, Rausher M. 2004. Evolution of mixed strategies of plant defense allocation against natural enemises. Evolution, Vol 58:1685-1695.
7. Boller T. 1995. Chemoperception of microbial signals in plant cells. Annual Review Plant Physiology Plant Molecular Biology, vol 46:189-214.
8. Yang YO, et al. 1997. Signal perception and transduction in defense responses. Genes development, vol 11. 1621-1639
9. Halverson LJ, Stacey G.1986. Signal exchange in plant-microbe interactions. Microbiology Reviews. Vol, 50: 193-225.
10. Ebel J, Cosio EG. 1994. Elicitors of plant defense responses. International Review Cytology, Vol148, 1-36.
11. Bonas U, Lahaye T. 2002. Plant disease resistance triggered by pathogen-derived molecules: refined models of specific recognition. Current Opinion Microbiology, Vol 5:44-50.
12. Van den Ackerveken G, Bonas U. 1997. Bacterial avirulence proteins as triggers of plant disease resistance. Trends Microbiology, Vol 5:394-398.
13. Flor HH. 1956. The complementary genetic system in flax and flax rust. Advance Genetics, Vol 8: 29-54
14. Dangl Jl, Jones J. 2001. Plant pathogens and integrated defence responses to infection. Nature, Vol 411: 826-833.
15. Logemann E, Wu SC, Schroder J, Schmelzer E, et al. 1995. Gene activation by UV light, fungal elicitor or fungal infection in Petroselium crispum is correlated with repression of cell cycle-related genes. Plant Journal, Vol 8: 865-876.
16. Alvarez ME. 2000. Salicylic acid in the machinery of hypersensitive cell death and disease resistance. Plant Molecular Biology, Vol 44: 429-442.
17. Hammond-Kosack KE, Parker JE. 2003. Deciphering plant-pathogen communication: fresh perspectives for molecular resistance breeding. Current Opinion Biotechnology, Vol 14:177-193.
18. Glazebrook J. 1996. Isolation of Arabidopsis mutants with enhanced disease susceptibility by direct screening. Genetics 143:973-982.
19. Glazebrook J. Rogers E. Ausubel F. 1997. Use of Arabidopsis for genetic dissection of plant defense responses. Annual Review Genetics, Vol 31: 547-569.
20. Parker JE. 2003. Plant recognition of microbial patterns.Trends Plant Sciences, Vol 8:245-247.
21. Gomez-Gomez L. 2004. Plant perception systems for pathogen recognition and defence. Molecular Immunology, Vol41:1055-1062.
22. Parker JE, Coleman MJ. 1997. Molecular intimacy between proteins specifying plant-pathogen recognition. Trends Biochemical Science, Vol 22:291-296.
23. Nurnberger T, Wirtz W, Nennstiel D, Hahlbrock K, et al. 1997. Signal perception and intracellular signal transduction in plant pathogen defense. Journal Receptor Signal Transduction Research, Vol 17:127-136.
24. Guttman D. 2004. Plants as models for the study of human pathogenesis. Biotechnology Advances, Vol 22: 363-382.
25. Gómez-Gómez L, Felix G, Boller T. 1999. A single locus determines sensitivity to bacterial flagellin in Arabidopsis thaliana. Plant Journal, Vol 18: 277-284.
26. Gómez-Gómez L, Boller T. 2000. FLS2: A LRR receptor-like kinase involved in recognition of the flagellin elicitor in Arabidopsis. Molecular Cell, Vol 5: 1003-1011.
27. Smith KD, Ozinsky A. 2002. Toll-like receptor-5 and the innate immune response to bacterial flagellin. Current Top Microbiology Immunology, Vol 270:93-108.
28. Imler JL, Ferrandon D, Royet J, Reichhart JM, et al. 2004. Toll-dependent and Toll-independent immune responses in Drosophila. Journal Endotoxin Research, Vol 10:241-246.
29. Felix G, Duran JD, Volko S, Boller T. 1999. Plants have a sensitive perception system for the most conserved domain of bacterial flagellin.
Plant Journal, Vol 18:265-276.
30. Gómez-Gómez L, Bauer Z, Boller T. 2001. Both the extracellular leucine-rich repeat domain and the kinase activity of FLS2 are required for flagellin binding and signaling in Arabidopsis. Plant Cell, Vol 13:1155-1163.
31. Gomez-Gomez L, Boller T. 2002. Flagellin perception: a paradigm for innate immunity. Trends Plant Science, Vol 7:251-256.
32. Castro MS, Fontes W. 2005. Plant defense and antimicrobial peptides. Protein Peptides Letters, Vol 12:13-18.
33. McDowell J, Woffenden B. 2003. Plant disease resistance genes: recent insights and potencial applications. Trends in Biotechnology, Vol 4:178-183.
34. Van der Biezen EA, Jones JDG. 1998. Plant disease-resistance proteins and the gene-for-gene concept. Trends Biochemistry Science, Vol 12: 454-456.
35. Martin GB, Brommonschenkel SH, Chunwongse J, Frary A, et al. 1993. Map-based cloning of a protein kinase gene conferring disease resistance in tomato. Science, Vol 262:1432-1436.
36. Pedley KF, Martin GB. 2003. Molecular basis of Pto-mediated resistance to bacterial speck disease in tomato. Annu Review Phytopathology, Vol 41:215-243.
37. Bonas U, Van den Ackerveken G. 1997. Recognition of bacterial avirulence proteins occurs inside the plant cell: a general phenomenon in resistance to bacterial disease?. Plant Journal, Vol 12:1-7.
38. Salmeron JM, Oldroyd GE, Rommens CM, Scofield SR, et al. 1996. Tomato Prf is a member of the leucine-rich repeat class of plant disease resistance genes and lies embedded within the Pto kinase gene cluster.
Cell, Vol 86:123-133.
39. Wu A-j, Andriotis V, Durrant M, Rathjen J. 2004. A match of surface-exposed residues mediates negativa regulation of immune signaling by tomato Pto kinase. The Plant Cell, Vol 16:2809-2821.