En organismos eucariotas operan una serie de mecanismos que se engloban bajo el término de ‘silenciamiento por RNA' y cuya base está en el reconocimiento específico de secuencias por pequeñas moléculas de RNA. El gran avance experimentado en este campo en los últimos años ha revelado un papel fundamental del silenciamiento por RNA en la regulación de la expresión de ciertos genes, en la defensa frente a virus y en el control de la estabilidad del genoma.
Los primeros indicios de la existencia de mecanismos de silenciamiento génico se obtuvieron a principios de los años 90, a partir de estudios con plantas transgénicas en las que se trataba de sobreexpresar un gen introduciendo copias extra mediante transformación génica. En muchos de estos casos los resultados obtenidos fueron contrarios a lo esperado: en lugar de una mayor expresión del producto génico, se producía una anulación de su expresión, provocada por una degradación específica de los RNA mensajeros (1). Un proceso similar al observado en plantas transgénicas también se observó en hongos. Sin embargo, el gran avance en este campo se produjo a finales de los 90, cuando se determinó que procesos similares al silenciamiento génico observado en plantas operan de forma generalizada en muchos otros organismos eucariotas (2, 3). Esto permitió un avance muy rápido en la caracterización bioquímica de estos procesos, englobados bajo el término de silenciamiento por RNA, y en la comprensión del papel que juegan en los organismos eucariotas (4).
A nivel molecular, el silenciamiento por RNA comienza en la célula a partir de la formación de RNA de doble cadena o dsRNA, que puede originarse de diversas maneras (Figura 1). El RNA de doble cadena es procesado por una enzima de tipo RNasa III, específica de dsRNA, denominada Dicer, que da lugar a fragmentos de RNA de entre 21 y 26 nucleótidos aproximadamente, denominados RNAs pequeños interferentes o siRNAs. Estos siRNAs son el común denominador de este tipo de procesos, al servir de guía a complejos proteicos efectores (RISC) que promueven la degradación de RNAs, inhibición de la traducción o metilación de DNAs que contengan secuencias complementarias al siRNA (5, 6).
A través de procesos de este tipo se regula la expresión de multitud de genes en una gran variedad de organismos eucariotas en momentos clave del desarrollo (7). En algunos organismos como plantas e insectos, el silenciamiento por RNA sirve además como defensa frente a virus, ya que la mayoría producen en algún momento de la infección RNAs de doble cadena que son reconocidos por la maquinaria celular y disparan una respuesta de degradación del RNA viral. Otro papel fundamental del silenciamiento por RNA es el control de elementos transponibles, donde parece tener gran relevancia la metilación de estas secuencias de DNA en el genoma y la consecuente inhibición de la transcripción de estos elementos (8).
La relevancia de cada una de las funciones del silenciamiento por RNA expuestas anteriormente, es variable entre unos organismos y otros (8). La regulación de la expresión génica y el control de elementos transponibles a través de procesos de silenciamiento por RNA podrían funcionar en multitud de organismos eucariotas. Sin embargo la defensa frente a virus parece tener mayor relevancia en plantas y en insectos. En estos, además, las rutas de silenciamiento por RNA parecen haberse diversificado y especializado al respecto, de acuerdo con la función que cumplen. La mosca Drosophila melanogaster contiene dos tipos de proteínas Dicer, y la planta Arabidopsis thaliana contiene cuatro: una de ellas, denominada DCL1, produce un tipo de pequeños RNAs (micro-RNAs) implicados en la regulación de la expresión génica, DCL2 participa en la defensa frente a virus, DCL3 es la encargada de producir siRNAs que dirigen la metilación de DNA y por último DCL4 está implicada en la producción de un subtipo de pequeños RNAs denominados trans-acting siRNAs (9, 10). Una situación similar existe con las proteínas de tipo Argonauta (AGO) que forman parte del complejo RISC efector del silenciamiento, y de las que se conocen varios tipos, cada una de las cuales podría participar en una ruta distinta de silenciamiento por RNA. Esta diversificación y especialización de las rutas de silenciamiento por RNA podría suponer una ventaja, al permitir al organismo regular de manera distinta unas rutas y otras. Por ejemplo en caso de infección viral el organismo podría potenciar la ruta de silenciamiento involucrada en la defensa antiviral, sin necesidad de alterar otras rutas de silenciamiento implicadas en otros procesos, como la regulación de la expresión génica.
Amplificación y diseminación sistémica del silenciamiento
Al núcleo del mecanismo de silenciamiento por RNA, representado por el procesamiento de dsRNA por Dicer y el efecto sobre RNAs diana mediado por RISC, se suman otros elementos que solo parecen estar presentes en algunos organismos, como son la amplificación y la diseminación del silenciamiento.
En plantas, hongos y nemátodos, los siRNAs además de promover la degradación de RNAs diana también son capaces de promover la formación de dsRNA de novo a partir de estos RNAs diana. En este proceso intervienen enzimas RNA polimerasas dependientes de RNA (RdRP ó RDR) (Figura 1), que sintetizan una cadena complementaria al RNA diana, posiblemente usando siRNAs como iniciadores de la reacción (11). A partir de este dsRNA se forma un nuevo conjunto de siRNAs lo que conlleva una amplificación de la respuesta de silenciamiento por RNA. Este mecanismo de amplificación parece tener gran importancia en el control de elementos transponibles y en la defensa frente a ciertos virus en plantas, ya que en plantas con mutaciones en algunos genes de enzimas RDR estas funciones se ven alteradas (9). La amplificación permite al organismo establecer procesos de silenciamiento incluso cuando la cantidad de dsRNA inicial es muy pequeña.
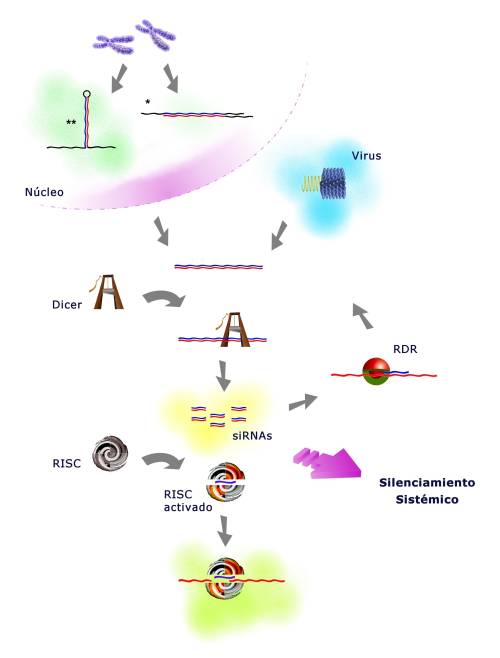 |
Figura 1 : El mecanismo de silenciamiento por RNA se inicia a partir de la producción de RNA de doble cadena (*) o RNA con estructura secundaria (**), que puede originarse a partir de la transcripción en el núcleo, durante infecciones virales o por la acción de RNA polimerasas (RDR) que lo sintetizan a partir de RNA de cadena simple. El RNA de doble cadena es procesado por enzimas Dicer para formar RNAs pequeños interferentes (siRNAs). Estos se incorporan a complejos proteicos (RISC) guiando su actividad, que suele conllevar degradación de RNAs diana. Es posible además que los siRNAs guíen la actividad de enzimas RDR en la producción de nuevo dsRNA, amplificando la respuesta de silenciamiento, o que se trasladen a células circundantes provocando el silenciamiento sistémico. |
Otro proceso aparentemente exclusivo de plantas y nemátodos es el silenciamiento sistémico. En estos organismos, la inducción de silenciamiento en una zona puede generar una señal móvil que inicie el silenciamiento en otras zonas distantes del organismo. Esta señal podría estar constituida por siRNAs que transiten de unas células a otras. En plantas, los siRNAs generados en una célula son capaces de pasar a células adyacentes a través de los plasmodesmos y mediar silenciamiento en ellas. Además, si hay RNAs diana en estas células receptoras se puede producir amplificación, lo que provocaría una mayor diseminación del silenciamiento (12). También es posible el tránsito de siRNAs a través del floema, lo que permitiría el movimiento del silenciamiento desde la zona donde se induce inicialmente hasta otras partes muy distantes en la planta (13, 14). Este movimiento del silenciamiento podría tener una gran importancia en la defensa frente a virus, ya que a partir del sitio inicial de infección, donde se induce el silenciamiento, se produciría una señal que prepararía la defensa antiviral en los tejidos adyacentes, antes de la llegada del virus, imponiendo una barrera a su dispersión por el organismo.
Como se ha comentado con anterioridad, el silenciamiento por RNA se inicia a partir de RNA de doble cadena, que puede tener diversos orígenes. En organismos eucariotas existen genes, descubiertos recientemente, que no codifican proteínas sino RNAs altamente estructurados, con porciones de doble cadena, que son procesados por enzimas de tipo Dicer para dar lugar a RNAs de 21-26 nucleótidos denominados micro-RNAs (miRNAs) (15). Estos miRNAs comparten muchas características con los siRNAs convencionales pero, a diferencia de estos, cada RNA precursor suele dar lugar a un solo miRNA. Cada miRNA se incorpora a un complejo RISC donde induce su acción. Los miRNAs tienen secuencias complementarias a la de RNAs mensajeros de diversos genes y, según el grado de complementariedad (16), pueden promover el corte del RNA diana o la inhibición de su traducción. En plantas los miRNA habitualmente aparean dentro de la región codificante de los RNAs diana con un alto grado de complementariedad, y mayoritariamente promueven el corte y posterior degradación de los RNAs diana. En cambio en animales predomina la inhibición traduccional y los miRNAs suelen aparear con un menor grado de complementariedad en múltiples puntos de la región 3' no traducible de los RNAs diana (4, 7).
Los genes descritos cuya expresión está regulada por miRNAs son con frecuencia factores de transcripción y genes implicados en el desarrollo (8). En plantas de A. thaliana, con alteraciones en genes implicados en la síntesis y función de miRNAs, aparecen graves alteraciones morfológicas y del desarrollo, lo que indica la importancia de la regulación de genes por miRNAs. Con el descubrimiento de nuevos miRNAs se está llegando a la conclusión de que estas moléculas podrían estar implicadas en otros procesos celulares además de la regulación del desarrollo, como mecanismos de homeostasis celular o incluso en la defensa frente a virus (17). En humanos se ha estimado que la regulación por miRNAs podría afectar hasta a un tercio del total de genes, lo que indica la gran relevancia de este tipo de mecanismos en el funcionamiento celular (18).
La regulación de genes mediante miRNAs podría tener varias ventajas respecto a otros mecanismos de regulación génica. La regulación a la baja de la expresión de un gen a través de miRNAs es probablemente mucho más rápida que la regulación mediante una disminución en su transcripción, lo que facilitaría cambios rápidos en el metabolismo o cambios muy localizados en tejidos concretos. Un único miRNA puede además regular de la misma manera a varios genes de una misma o distinta ruta metabólica, siempre que estos contengan secuencias complementarias al miRNA, lo que facilita la coordinación en los cambios de expresión de tales genes. Además, permiten una fácil regulación mediante retroalimentación, si los genes diana están implicados en la síntesis o función de los miRNAs. Así por ejemplo, los RNA mensajeros de DCL1 y de AGO1, genes implicados en la síntesis y en la función de miRNAs en plantas, están a su vez regulados por miRNAs (19).
Una de las funciones del silenciamiento por RNA es el control de la actividad de elementos transponibles, a través de la formación de heterocromatina. Los elementos transponibles, transposones y retrotransposones, son secuencias de DNA muy abundantes en el genoma de eucariotas que tienen la capacidad de moverse de un punto a otro en el genoma (20). La actividad de estos elementos conlleva la posibilidad de que se produzcan mutaciones perniciosas, por ejemplo al insertarse un elemento transponible dentro de un gen, provocando la pérdida de su funcionalidad. La actividad potencialmente peligrosa de estos elementos transponibles es controlada a través de la formación de heterocromatina. La heterocromatina es DNA altamente condensado y básicamente inactivo, la mayoría de la cual se encuentra en el centrómero y en los telómeros de los cromosomas, donde abundan secuencias de DNA altamente repetitivas. La formación de heterocromatina conlleva modificaciones como la metilación del DNA y de ciertos aminoácidos de las histonas. En este proceso, el silenciamiento por RNA juega un papel relevante, según se ha puesto de manifiesto por la abundancia en diversos organismos eucariotas de siRNAs correspondientes a secuencias de elementos transponibles y de DNA repetitivo y, además, por el descubrimiento de que los siRNAs son capaces de provocar la metilación del DNA con el que tienen homología de secuencia. También se ha observado que la alteración de ciertos genes implicados en silenciamiento por RNA causa una reducción en la metilación del DNA y una mayor actividad de elementos transponibles, lo que implica a este mecanismo en el control de la actividad de estos elementos potencialmente dañinos para la estabilidad del genoma (21).
La inducción de silenciamiento por parte de estos elementos transponibles podría verse facilitada por el hecho de que están muy repetidos en el genoma. La inserción contigua de varios elementos, o incluso la inserción de uno dentro de otro, podría causar la producción de RNAs mensajeros con secuencias complementarias que formasen RNAs de doble cadena. Adicionalmente, la formación de transcritos de RNA conteniendo repeticiones o alguna otra peculiaridad, podría ser reconocida por enzimas RDR que sintetizasen un RNA complementario, formando así dsRNA. De hecho en A. thaliana una de estas enzimas, RDR2, juega un papel relevante en el silenciamiento de elementos transponibles (9).
Los virus son parásitos intracelulares capaces de transmitirse de un organismo huésped a otro y de utilizar la maquinaria celular del organismo al que infectan para replicarse (22). Los virus, además, suelen causar alteraciones en el metabolismo y desarrollo de los organismos a los que infectan que conllevan una pérdida de valor adaptativo de estos a su entorno. En plantas y otros organismos, una de las funciones del silenciamiento por RNA es la defensa frente a virus. Muchos virus producen en alguna fase de su ciclo de infección RNAs de doble cadena. A partir de estos dsRNAs se producen siRNAs con secuencias del virus que promueven la degradación de RNAs virales, lo que conduce a una limitación en la acumulación y dispersión del virus dentro del organismo huésped (23, 24).
Las posibles fuentes de producción de dsRNA a partir del virus son diversas. Los virus cuyo genoma está constituido por RNA, producen durante la replicación RNAs complementarios al RNA viral, que sirven de molde para la síntesis de nuevos RNAs virales. Estos RNAs de ambas polaridades forman de manera transitoria dsRNAs, denominados intermediarios replicativos, que probablemente son reconocidos por la célula como inductores de silenciamiento (25). Algunos virus producen durante la infección mayor cantidad de siRNAs correspondientes a una zona concreta de su genoma, por lo que se cree que podrían inducir silenciamiento a partir de estructuras secundarias en el RNA viral que contengan porciones de dsRNA. Otros virus, cuyo genoma está constituido por DNA, pueden formar dsRNA durante la transcripción de sus RNA mensajeros. Es posible incluso que se produzcan dsRNAs a partir de la acción de enzimas RDR del organismo huésped sobre el RNA viral, ya que la alteración de algunas de estas enzimas en A. thaliana provoca una mayor susceptibilidad a ciertos virus. Los viroides, constituidos únicamente por un RNA circular que no codifica proteínas, podrían inducir silenciamiento debido a la síntesis de un RNA complementario al RNA genómico durante su replicación, o a partir de porciones de dsRNA que se formarían en zonas autocomplementarias de su RNA. Al igual que las posibles fuentes de dsRNA son variables dependiendo del tipo de virus, las rutas de silenciamiento por RNA que intervienen en la defensa antiviral podrían también ser distintas para cada tipo de virus. Así pues, en A. thaliana la enzima DCL2 parece estar implicada en la defensa frente a unos tipos de virus pero no frente a otros. Igualmente, las enzimas RDR1 y RDR6 parecen estar implicadas en la defensa frente a distintos tipos de virus. Por ello, es posible que la diversificación de rutas de silenciamiento por RNA, observada en algunos organismos como plantas, tenga como objetivo una mejor adaptación de la defensa antiviral frente a distintos tipos de virus (8, 25). La defensa frente a virus basada en silenciamiento por RNA tiene como principal ventaja su adaptabilidad, ya que la célula reconoce un aspecto intrínseco a la infección viral, como es la producción de dsRNA, e inicia una defensa cuya especificidad viene determinada por la propia secuencia de nucleótidos del virus.
A pesar del papel que juega el silenciamiento por RNA como defensa antiviral es evidente que los virus, al menos en algunas situaciones, son capaces de infectar sus plantas huéspedes. Para ello, los virus han desarrollado mecanismos de contradefensa frente al silenciamiento por RNA. Algunos de estos mecanismos tratan de eludir el silenciamiento, por ejemplo llevando a cabo la replicación viral en vesículas membranosas, aisladas de la maquinaria de silenciamiento. Otros virus mutan muy rápidamente durante la infección, con lo que el silenciamiento ve reducida su capacidad de hacer diana en el RNA viral. Algunos virus y viroides son resistentes a la degradación por silenciamiento, posiblemente porque la estructura secundaria de sus RNAs o su interacción con proteínas dificulta la accesibilidad del complejo RISC. Por último, la rápida replicación y dispersión de algunos virus de unas células a otras también podría tener como objetivo evadir el silenciamiento (25).
Algunos virus van más allá de la mera evasión de la defensa antiviral y son capaces de suprimir activamente el mecanismo de silenciamiento por RNA a través de proteínas codificadas en su genoma. Se han identificado un gran número de proteínas, mayoritariamente de virus de plantas, capaces de suprimir silenciamiento (25, 28, 32). Estas proteínas tienen orígenes evolutivos distintos, lo que indica que muchos virus han convergido evolutivamente en la estrategia de suprimir el silenciamiento por RNA. Además, estas proteínas suelen ser factores de patogenicidad necesarios para la correcta acumulación y propagación del virus, lo que confirma la necesidad del virus de suprimir el silenciamiento por RNA para poder infectar a su huésped. Los modos de acción de estas proteínas son muy diversos. Algunas son capaces de interferir directamente con componentes de la maquinaria de silenciamiento, como es el caso de la proteína p19 de tombusvirus y la p21 de closterovirus, que son capaces de unirse a siRNAs, posiblemente impidiendo su incorporación al complejo RISC. Las proteínas NS1 del virus de la gripe y E3L del virus vacunal son capaces de suprimir silenciamiento posiblemente debido a su capacidad de unirse a dsRNA (25). Otras proteínas supresoras podrían mediar su acción interaccionando con proteínas de la planta reguladoras del silenciamiento. Así por ejemplo, la proteína HC-Pro de potyvirus interacciona con una proteína de la planta de tipo calmodulina que tiene capacidad inhibidora del silenciamiento (26). Otro posible modo de acción es actuar directamente sobre la expresión de ciertos genes de la planta. Las proteínas TrAP/AC2 de geminivirus actúan como activadores de la transcripción de varios genes de la planta, entre ellos genes cuya sobreexpresión podría interferir con el silenciamiento por RNA (25, 27).
Las consecuencias para el mecanismo de silenciamiento por RNA varían dependiendo del modo de acción del supresor en cuestión (28). La mayoría son capaces de impedir o reducir la acción del silenciamiento en el propio tejido donde se expresa el supresor. Otras, como la p25 de potexvirus y 2b de cucumovirus, no impiden el silenciamiento en el propio tejido, sino que interfieren con la producción o exportación de la señal que inicia el silenciamiento en órganos adyacentes, lo que podría favorecer la dispersión del virus (29, 30). El modo de acción de la proteína supresora determina también que la supresión se limite a las rutas de silenciamiento implicadas en defensa antiviral o afecte también a otras rutas. De manera destacable, algunos supresores son capaces de interferir con las rutas de regulación de genes mediadas por miRNAs. La interferencia con estas rutas, observada en plantas, provoca serias alteraciones en el desarrollo, similares a las causadas por mutaciones en genes que participan en la producción o actividad de miRNAs. Esta interferencia con el metabolismo de miRNAs es por tanto una de las causas de los síntomas producidos por algunos virus en plantas (31). Actualmente, no se conoce si la interferencia de estas proteínas virales con el metabolismo de miRNAs es un efecto secundario de la supresión de la defensa antiviral o si por lo contrario el virus obtiene alguna ventaja de ello, al variar el patrón de expresión de algunos genes de la planta de manera beneficiosa para el propio virus.
Los virus, como parásitos obligados, dependen del organismo huésped para sobrevivir. Es por tanto factible pensar que la evolución de los virus vaya dirigida a causar el mínimo daño al organismo huésped, lo que redundaría en una mayor supervivencia del propio virus. Algunos casos observados en la naturaleza demuestran que una excesiva acumulación del virus puede ser muy perjudicial para el organismo huésped, por lo que es probable que la supresión de la defensa del huésped mediada por silenciamiento por parte de los virus, se haya adaptado en sistemas evolutivos avanzados hasta alcanzar un equilibrio entre silenciamiento y supresión de este, que promueva una óptima acumulación del virus (32). En plantas, algunos virus suprimen muy poco la respuesta de defensa basada en silenciamiento, lo que provoca una acumulación muy baja del virus que causa muy pocas alteraciones a la planta. Esto puede ser beneficioso para estos virus ya que, por ejemplo, las plantas infectadas pueden florecer normalmente, lo que permite la dispersión del virus a través del polen.
Otra forma en que los virus podrían sacar provecho del silenciamiento consiste en redirigir este proceso hacia genes del organismo huésped. Entre los virus de plantas es posible encontrar secuencias con cierto grado de similitud con genes de plantas. Esto sugiere que una vez iniciado el silenciamiento frente al virus, algunos siRNAs producidos a partir del dsRNA viral podrían tener suficiente identidad de secuencia con ciertos RNA mensajeros de la planta como para dirigir su degradación (25, 33). A favor de esta hipótesis, se ha observado que la expresión en plantas de un dsRNA que porta secuencias de un viroide sin capacidad replicativa, es capaz de causar síntomas similares a los observados en una infección con el viroide. De esta manera, es posible que los virus alteren la expresión de genes del organismo huésped en su propio beneficio, por ejemplo promoviendo la degradación de RNA mensajeros de genes implicados en la defensa. Igualmente, la infección viral podría causar una saturación de la maquinaria de silenciamiento, causando una sobreexpresión de genes que normalmente se encuentren silenciados.
El silenciamiento por RNA puede ser utilizado como herramienta con diversos fines. Una de las posibles aplicaciones es el estudio de la función de genes, evaluando el efecto que tiene sobre el organismo la eliminación del producto génico mediante la expresión de dsRNA que porte secuencias del gen, lo que se conoce como interferencia por RNA o RNAi (34). La expresión de dsRNA se puede producir de manera estable, mediante transformación genética con una construcción que exprese un RNA con secuencias sentido y antisentido. En algunos organismos como nemátodos y embriones de Drosophila , se puede inducir RNAi mediante la introducción directa de dsRNA sintetizado in vitro , aunque el efecto logrado sobre el gen diana es transitorio. En mamíferos, la inducción de RNAi mediante expresión de dsRNA no es viable, ya que el dsRNA activa enzimas proteín-quinasas que promueven otras respuestas inespecíficas que conllevan muerte celular. Sin embargo, se puede inducir RNAi mediante la expresión o introducción directa de siRNAs que no activan las respuestas mediadas por proteín-quinasas.
En plantas, además de la expresión estable de dsRNA mediante transgenes, se pueden utilizar a los virus como herramientas para el estudio de la función de genes (35). Modificando los virus para que porten secuencias del gen de interés, se logra que el silenciamiento inducido por el virus durante la infección tenga como diana el gen de la planta. Este procedimiento se conoce como silenciamiento inducido por virus o VIGS. Algunas de sus ventajas son la facilidad de uso, ya que es mucho menos laborioso que la transformación genética con construcciones que expresen dsRNA, y la posibilidad de controlar a partir de que momento se induce el silenciamiento, lo que permite por ejemplo trabajar con genes implicados en el desarrollo, que harían a la planta inviable en caso de ser silenciados en un estadio temprano. Como inconveniente del VIGS está la posibilidad de que los síntomas causados por la infección viral enmascaren el fenotipo causado por el silenciamiento del gen de interés.
Otra de las aplicaciones del silenciamiento por RNA es la creación de plantas transgénicas resistentes a virus, mediante la transformación con construcciones que expresen dsRNA con secuencias del virus de interés. De esta manera, se pueden reducir las pérdidas ocasionadas por ciertas enfermedades virales en plantas de cultivo. Las nuevas tendencias van incluso hacia la protección de cultivos sin necesidad de utilizar plantas transgénicas, mediante la aplicación directa de dsRNAs, utilizando métodos que faciliten la entrada de este a las células (36). La utilización del silenciamiento por RNA con similares fines en humanos, topa con la dificultad de dirigir in vivo las moléculas efectoras hacia las células diana (34). Una posibilidad es utilizar vectores virales que expresen las secuencias de interés en las células diana, lo que conlleva un riesgo biológico por el empleo de material infectivo en pacientes. Otra posibilidad es manipular células del paciente in vitro , introduciendo secuencias inductoras del silenciamiento, y proceder después a la reimplantación de las células tratadas en el paciente.
El silenciamiento por RNA surgió como un inconveniente fortuito para la expresión de transgenes en plantas. Sin embargo, tras comprender su papel natural se ha revelado como un complejo sistema de regulación de la expresión génica en multitud de organismos eucariotas. Su presencia en la variedad de organismos eucariotas documentada sugiere un origen evolutivo remoto, antes de la diferenciación de eucariotas en grandes reinos. A la vez, su conservación en organismos tan dispares sugiere que pueda cumplir funciones de gran importancia biológica. En un futuro próximo es de esperar que se conozca con más detalle el modo de acción y el papel en el organismo de múltiples procesos relacionados con el silenciamiento por RNA. También, gracias a la posibilidad de dirigir el mecanismo de silenciamiento frente a dianas concretas con una alta especificidad, es de esperar que surjan nuevas terapias frente a diversas enfermedades en humanos y en otros organismos.