La aparición de miles de marcos de lectura abiertos que codifican nuevas proteínas, como consecuencia del éxito en la secuenciación de diferentes genomas, ha planteado el gran reto de obtener información sobre la función y localización celular de las mismas. Una estrategia desarrollada en los últimos años consiste en unir estas secuencias a la de proteínas fluorescentes (FPs, fluorescent proteins). Las FPs generan fluorescencia espontáneamente al ser sintetizadas por un sinfín de tipos celulares y pueden estudiarse en células vivas sin necesidad de fijación y tinción. Estas proteínas están revolucionando la biología celular al permitir obtener datos sobre localización, dinámica y redes de interacción molecular de numerosas proteínas con una gran resolución temporal y espacial 1 . A partir de la forma silvestre de la proteína fluorescente verde (GFP, green fluorescent protein ) de la medusa A. victoria (y más recientemente de proteínas similares derivadas de ciertos corales) se han obtenido numerosas variantes en el laboratorio mediante mutagénesis, con el objeto de mejorar sus propiedades ópticas y generar diversos colores 2 . Todo ello ha permitido usar técnicas clásicas de microscopía de fluorescencia multicolor en células vivas, así como otras técnicas más sofisticadas que suelen designarse con las siglas inglesas, como FRAP (recuperación de fluorescencia tras fotodestrucción), FCS (espectroscopía de correlación de fluorescencia) y FRET (transferencia de energía resonante de fluorescencia).
Las FPs, además de servir como marcadores de localización o interacción molecular, se usan para monitorizar cambios bioquímicos que ocurren en el interior de células vivas mediante sondas moleculares recombinantes. Cuando se unen a iones, segundos mensajeros, o sufren modificaciones como fosforilación, proporcionan una señal fluorescente. Al tratarse de fragmentos de ADN fusionados linealmente a voluntad, permiten la construcción de organismos genéticamente modificados que expresan el transgén en los tejidos que desee el investigador.
En este artículo se revisarán los aspectos más relevantes de las FPs con énfasis en algunas de sus aplicaciones en Biología Celular y Biotecnología, las cuales se resumen en la Tabla 1.
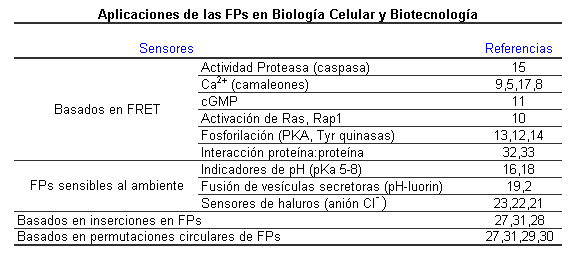 |
Tabla 1 |
La GFP, descubierta en 1962, acompaña a la fotoproteína ecuorina de la medusa Aequorea victoria y convierte la luminiscencia azul de la ecuorina en fluorescencia verde, mediante un proceso de transferencia de energía sin emisión y reabsorción de radiación. En 1992 se determinó la secuencia del cDNA de GFP Aequorea y se demostró que su expresión heteróloga en organismos distintos de la medusa produce fluorescencia sin requerir cofactor alguno. La GFP es una proteína de 238 aa y por lo tanto, cuando se utiliza en una proteína de fusión no es un marcador pequeño. Tiene mayor tamaño que las etiquetas químicas que se usan para marcar proteínas (FITC, TRITC), pero menor que los anticuerpos utilizados en inmunocitoquímica. Su principal ventaja consiste en que la fluorescencia se genera espontáneamente en células vivas. Se ha identificado la estructura química de su fluorocromo, 4-(p-hidroxibencilidina)imidazolidina-5-ona, el cual se forma a partir de los residuos 65-67 (Ser-Tyr-Gly) de la GFP mediante una modificación post-traducción en tres reacciones 1 . Durante la formación del grupo fluorescente se forma H 2 O 2 como producto secundario de la oxidación de GFP por el O 2 . La Gly67 se conserva en todos los mutantes de GFP conocidos que retienen fluorescencia.
La GFP se cristalizó por primera vez en 1974, pero no se pudo determinar su estructura cristalina hasta 1996 3,4 . Es un barril formado por 11 hojas plegadas beta atravesado por una hélice alfa coaxial, donde se halla el cromóforo. La GFP Aequorea tiene cierta tendencia a dimerizar mediante interacciones hidrofóbicas e hidrofílicas. De hecho, la estructura cristalina publicada por el grupo de Phillips es un dímero y esto puede limitar algunas aplicaciones biotecnológicas. Por ejemplo, si se quisiera estudiar la oligomerización de una proteína concreta, no sería apropiado utilizar como marcador fluorescente otra proteína que tenga tendencia a dimerizar por sí misma. Sin embargo, como los residuos que forman la interfase entre los dos monómeros de GFP son conocidos, se han eliminado mediante mutagénesis para minimizar esta tendencia.
La Tabla 2 resume las propiedades de las principales familias de GFP mutadas: GFP silvestre (con dos picos de excitación a 396 y 472 nm), GFP con anión fenolato (S65T, RSGFP4, EGFP), GFP con fenol neutro (T203I, sapphire ), GFP con anillo aromático junto al anión fenolato (10C, EYFP), GFP con indol en el cromóforo (ciano o ECFP), GFP con imidazol en el cromóforo (EBFP), y finalmente, las FPs rojas derivadas del coral Discosoma sp (DsRed, mRFP1). De ellas, las más usadas son la EGFP, ECFP y EYFP, y la DsRed, aunque esta última es un tetrámero obligado, con las limitaciones que hemos descrito en el párrafo anterior para los dímeros.
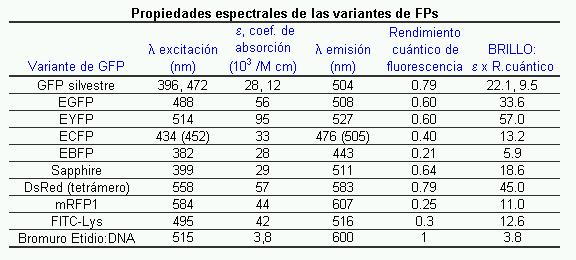 |
Tabla 2 |
En estudios de microscopía de fluorescencia multicolor, con frecuencia se transfectan células simultáneamente con dos FPs, por ejemplo, ECFP y EYFP, y a menudo interesa conocer la concentración relativa de ambas. Pues bien, de la Tabla 2 se puede deducir que dos células que presenten la misma intensidad de fluorescencia de ECFP y EYFP no tendrían la misma concentración de cada proteína y que la concentración de ECFP sería mayor que la de EYFP. El brillo de una FP, como para cualquier molécula fluorescente, depende del producto de ABSORCIÓN x FLUORESCENCIA, esto es, del producto de su coeficiente de extinción molar (e) por su rendimiento cuántico de fluorescencia (R.C., la proporción de fotones emitidos por fotón absorbido). Así, en la Tabla 2 se indica que este producto es 57 para EYFP y sólo 13,2 para ECFP. Es decir, mol por mol, EYFP es 4,3 veces más brillante que ECFP. Hay enormes diferencias entre distintas FPs y algunas de ellas (EBFP) se han abandonado por presentar una fluorescencia apenas superior al fondo producido por la autofluorescencia de las células y del sistema óptico. Sin embargo, la mayoría de las FPs son mejores fluorocromos que un conjugado de fluoresceína (e x R. C. = 12,6) o que el complejo etidio:ADN (e x R. C. = 3,8).
Hay que recordar que la intensidad de fluorescencia de una célula transfectada depende de la concentración y propiedades de la FP que exprese, pero también del sistema óptico en su conjunto: la intensidad de la luz de excitación, cercanía de la longitud de onda de excitación al pico de absorción de la FP, ancho de banda y transmisión del filtro de excitación, transmisión del objetivo, ancho de banda y transmisión del filtro de emisión, y rendimiento cuántico del detector. Por ejemplo, algunas cámaras CCD científicas tienen mayor rendimiento cuántico a 550 nm que a 450 nm, lo que en nuestro ejemplo juega de nuevo a favor de mayor intensidad registrada de EYFP que de ECFP (a igual concentración).
Existen al menos tres estrategias para la construcción de indicadores fluorescentes recombinantes para iones (Ca 2+ , H + , Cl - ), segundos mensajeros (cGMP) y señalización celular (activación de Ras, fosforilación):
1. Construir una proteína de fusión situando un receptor entre dos FPs de distinto color, que sean capaces de hacer FRET dependiendo de la presencia del ligando del receptor (ej. los sensores de Ca 2+ llamados “camaleones”, sensores de activación de Ras “Raichu”, otros).
2. Hacer que la propia FP sea sensible a un ión mediante mutagénesis (ej. indicadores de pH).
3. Utilizar permutaciones circulares de FPs unidas a receptores (ej. los sensores de Ca 2+ llamados “pericams”).
La transferencia de energía por resonancia (se suelen usar las siglas del inglés FRET, fluorescence resonance energy transfer ) consiste en que un fluorocromo aceptor de energía situado a cierta distancia de una molécula excitada (donante de energía) puede causar apagamiento ( quenching ) de esta última (Fig 1). Así, se acorta la vida media del estado excitado del donante y aparece fluorescencia inducida en el aceptor (sin que éste absorba fotones de excitación). La velocidad de transferencia de energía k FRET depende críticamente de la distancia (R) entre los fluorocromos (k FRET =1/R 6 ) y de su orientación relativa. De este modo, para la mayoría de pares de fluorocromos, FRET ocurre a distancias similares a las de las interacciones moleculares (3 a 10 nm). La relación de la distancia y la orientación con k FRET permite obtener una señal óptica acoplada a posibles interacciones entre proteínas marcadas con fluorocromos, con una resolución espacial hasta 100 veces superior al límite físico en microscopía óptica (que es de alrededor de 200 nm en el plano x-y, dependiendo de la longitud de onda).
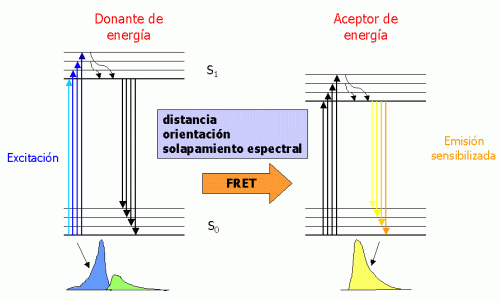 |
Figura 1: El estado excitado del fluorocromo donante de energía se relaja mediante la transferencia de energía (FRET) a otra molécula aceptora, la cual se excita sin absorber fotones y emite luz con su espectro característico. |
La eficiencia de FRET también depende de la diferencia de energía entre los fluorocromos donante y aceptor, lo que implica que k FRET es proporcional al solapamiento entre el espectro de emisión del donante y el de absorción del aceptor. Existe otro fenómeno que no debe confundirse con FRET: la absorción de fotones emitidos por el donante por parte del aceptor (que a su vez se excita y emite luz) es un fenómeno sin interés y poco útil, ya que no presenta la dependencia crítica con la distancia característica de FRET.
El primer tipo de indicadores recombinantes con FPs basados en FRET fueron los sensores para Ca 2+ que denominamos camaleones por contener calmodulina (CaM) y porque modifican su color de emisión fluorescente cuando la CaM se activa por Ca 2+ 5 . En esencia, en este tipo de construcciones dos FPs de distinto color flanquean un receptor apropiado (Fig 2). La unión del ligando al receptor produce un cambio conformacional que altera la distancia y/o orientación entre las FPs y resulta en un cambio de FRET. El camaleón contiene, además de CaM, un péptido de 27 aa (M13) derivado de la quinasa de la cadena ligera de la miosina (MLCK), el cual se une a la CaM de forma reversible en presencia de Ca 2+ (Fig 2). Al aumentar la concentración de Ca 2+ en células que expresan un camaleón aumenta la emisión verde-amarilla (EYFP, aceptor de energía) y disminuye la emisión azul (ECFP, donante de energía), todo ello iluminando las células con luz de 430 nm, que excita casi exclusivamente al donante ECFP (Fig 3). Este tipo de indicadores, a diferencia del fura-2 y demás sensores de síntesis, permitió medir cambios de Ca 2+ en algunas localizaciones específicas de la célula (núcleo, citoplasma, retículo endoplásmico) que hasta entonces sólo eran accesibles mediante la fotoproteína luminiscente ecuorina.
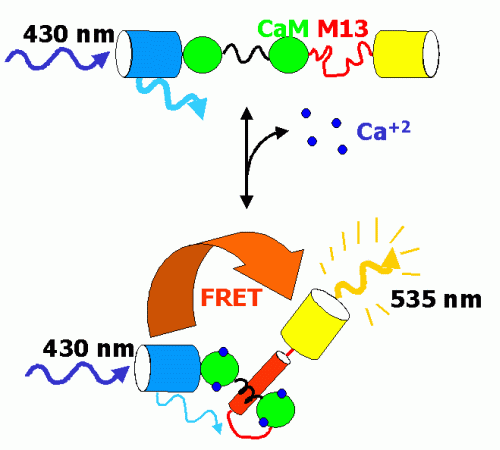 |
Figura 2: La unión reversible del Ca 2+ a la calmodulina (CaM) resulta en la unión al péptido M13 en una conformación más compacta. El cambio de distancia y orientación causado en las FPs aumenta la transferencia de energía (FRET) entre las mismas. |
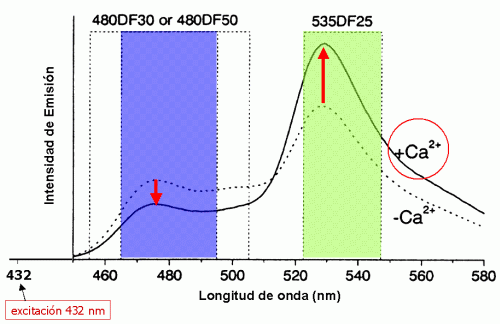 |
Figura 3: Espectros de emisión del sensor de Ca 2+ camaleón. En presencia de Ca 2+ aumenta la emisión amarilla (EYFP) y disminuye la azul (ECFP). |
El rango dinámico de los camaleones (su capacidad de detectar cambios de Ca 2+ en el rango de micromoles o milimoles) puede modularse introduciendo mutaciones en residuos específicos (Glu) de las llamadas “manos EF” de la CaM, que son los dominios responsables de unir Ca 2+ . Estas mutaciones disminuyen de forma predecible la afinidad CaM:Ca 2+ . De esta forma generamos camaleones de afinidad alta (K´d, constante de disociación 70 nM) y media (K´d 4,4 m M) útiles para medir Ca 2+ en el citoplasma y núcleo, mientras que para el retículo endoplásmico se utilizan camaleones de afinidad media (K´d 4,4 m M) y baja (K´d 700 m M). Hay que tener en cuenta que el retículo endoplásmico contiene una concentración de Ca 2+ muy superior al citoplasma, al tratarse de un depósito intracelular de este ión que se moviliza ante estímulos fisiológicos.
Por último, la ventaja más destacada de trabajar con indicadores fluorescentes recombinantes es la posibilidad de construir organismos transgénicos que expresan la quimera bajo el control de promotores específicos, y que permite abordar experimentos muy difíciles con otras técnicas. Por ejemplo, el estudio de la dinámica del Ca 2+ en plantas mediante indicadores de síntesis química (fura-2 y otros) está muy limitado por la baja difusión de los ésteres AM a través de la pared celular vegetal. Sin embargo, se han construido plantas transgénicas ( A. thaliana ) que expresan el camaleón en las células de los estomas 6 , así como nematodos transgénicos ( C. elegans ) que lo expresan en el músculo faríngeo 7 , y que han permitido estudiar detalles de la función del Ca 2+ que eran inaccesibles con los indicadores clásicos. Más recientemente se ha mejorado el rango dinámico de los camaleones sustituyendo el péptido M13 por otro péptido derivado de la quinasa dependiente de CaM-quinasa (CKKp) 8 . También se ha descrito una nueva familia de indicadores con troponina C como receptor de Ca 2+ (en lugar de CaM) que presentan mayor cambio de FRET y K´ds de 470 nM a 29 m M 9 .
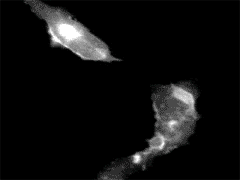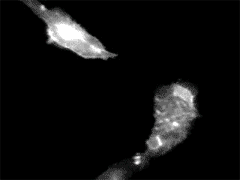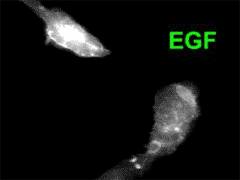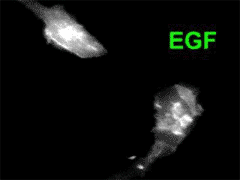
La figura 4 muestra otro indicador con un diseño muy similar al del camaleón, en el que se inspiraron sus autores. Se trata del sensor de activación de Ras denominado “Raichu Ras” 10 . Se ha sustituido la CaM por la proteína Ras, y el péptido M13 por un fragmento de Raf, una proteína activada por Ras-GTP. Cuando Ras se activa por mediación de un factor de intercambio de GTP (GEF, GTP exchange factor ), como SoS, aumenta la afinidad de Ras por Raf, la proteína quimérica cambia su conformación y aumenta la señal FRET entre ECFP y EYFP situadas en los extremos de la construcción (Fig 4). El cociente de emisión amarilla/azul aumenta con la activación de Ras, como ocurría con los camaleones al aumentar la concentración de Ca 2+ . También puede verse la actividad de la membrana plasmática ( ruffles ) ya que Raichu Ras está anclado a la misma (véase video).
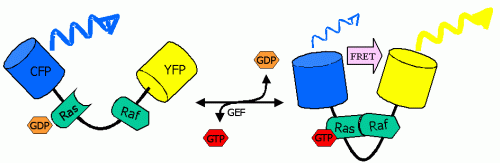 |
Figura 4: El sensor “Raichu Ras” responde a la activación de Ras con un aumento de la emisión amarilla (EYFP) y disminución de la azul (ECFP). |
 |
Los camaleones han servido de prototipo para otros indicadores basados en FRET: para detectar cGMP 11 (utilizando como receptor un fragmento de la proteína quinasa dependiente de cGMP); para detectar actividad tirosina quinasa 12,13 y proteína quinasa A 14 en células transfectadas con las quimeras; y como sensores de activación de caspasas 15 .
Varias de las FPs obtenidas mediante mutagénesis presentan una sensibilidad al pH muy superior a la de la GFP silvestre de A. victoria. Así, demostramos que EGFP presenta un pKa de 6 y EYFP de 7 16 (Fig 5). Esto implica que para EYFP, cualquier cambio de pH alrededor del nivel fisiológico (7 en el citoplasma) causará cambios sustanciales de la fluorescencia: aumenta a pH alcalino y disminuye a pH ácido. Cualquier proteína de fusión con FPs es potencialmente sensible a este artefacto, lo que hizo necesario buscar YFPs con pKas inferiores a 7 para que los camaleones fueran sensibles sólo a cambios de Ca 2+ y no a cambios de pH. Así surgieron las versiones de camaleón 2.1, 3.1 y 4.1 (de alta, media y baja afinidad, respectivamente), que incorporan YFP con las mutaciones V68L y Q69K, responsables de que el pKa sea 6, más alejado del valor fisiológico de pH citoplásmico. Estos camaleones mejorados permiten realizar medidas de Ca 2+ sin interferencia por una posible acidificación citosólica 17 .
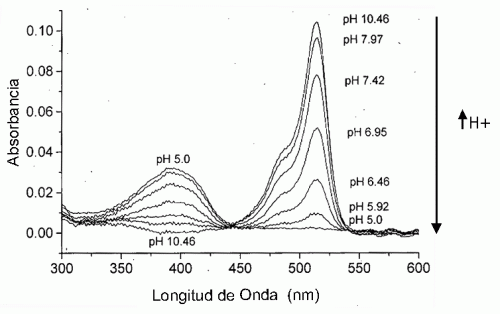 |
Figura 5: La proteína EYFP disminuye la absorción y la fluorescencia en ambiente acídico. |
La sensibilidad al pH de FPs mutadas, aunque resulta problemática para ciertas proteínas de fusión, puede ser útil en sí misma para construir sensores de pH cuya expresión se pueda dirigir específicamente al citoplasma, retículo endoplásmico, aparato de Golgi o matriz mitocondrial, entre otras localizaciones posibles. Demostramos la utilidad de esta estrategia en estudios de regulación del pH del aparato de Golgi 16 . Para el caso de la matriz mitocondrial, identificamos mutaciones que aumentan el pKa de la YFP a un valor de 8, cercano al pH de la matriz mitocondrial 18 , y dirigimos la expresión de esta variante mediante una secuencia de localización derivada de la enzima mitocondrial citocromo oxidasa.
Otra aplicación ingeniosa de la sensibilidad al pH de la YFP consiste en dirigir su expresión al interior de vesículas secretoras y sinápticas mediante fusiones con proteínas residentes en estas vesículas. De esta forma se ha conseguido medir procesos de secreción y transmisión sináptica 19,20 .
Por último, una discrepancia en el pKa calculado para YFP entre nuestros datos y los calculados por la Dra. Rebekka Wachter (Oregon State University) llevó al descubrimiento de que las YFPs presentan también sensibilidad a haluros, de los cuales el de mayor relevancia fisiológica es el ión cloruro 21 . De nuevo, esta propiedad, que hace que ciertas proteínas de fusión puedan variar su fluorescencia y causar problemas de interpretación ante cambios fisiológicos de niveles de Cl - intracelular, puede usarse en interés del investigador en la construcción de sensores útiles en estudios de canales y reguladores de Cl - , por ejemplo, CFTR en la fibrosis quística 22,23 .
Se ha descrito una variante de YFP con menor interferencia por pH y cloruro, la denominada “citrine” 24 . Otra mutación mejora, además, el paso limitante en la formación post-traducción del fluorocromo, resultando en la variante llamada “Venus” con una maduración acelerada a 37ºC 25,26 .
Durante un estudio de mutagénesis semirandomizada sobre ECFP se detectó la inserción casual de un hexapéptido (FKTRHN) en lugar de la Tyr145, y sorprendentemente la proteína seguía siendo fluorescente (Fig 6A) 27 . Esto hizo pensar que los dominios 1-145 y 146-238 (aproximadamente los dos tercios iniciales y el tercio final de ECFP) pueden plegarse y madurar independientemente. Para comprobar si esta hipótesis era correcta se hicieron inserciones de mayor tamaño y permutaciones circulares en FPs, lo que dio lugar a dos nuevas estrategias exitosas para la construcción de sensores basados en una sola FP (a diferencia de FRET, que requiere dos FPs).
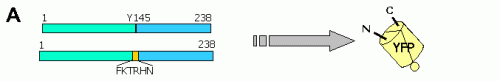 |
Figura 6A: La inserción de un hexapéptido en Tyr145 resultó en una quimera fluorescente |
La inserción de CaM en lugar de Tyr145 resultó en una quimera que aumentaba su emisión fluorescente con la concentración de Ca 2+ (con una K´d de 7 m M), y que se denominaron camgaroos (canguros, por analogía de los iones Ca 2+ saltando dentro y fuera de la bolsa marsupial que sería la CaM) (Fig 6B). Como sucedió con los camaleones, se han construido organismos transgénicos que expresan los camgaroos , en particular en ciertas neuronas del cerebro de la mosca D. melanogaster 28 . Si en lugar de CaM se insertaba un dedo de zinc ( Zn finger ), la proteína quimérica respondía de igual modo a la concentración ambiente de zinc, constituyendo un indicador fluorescente para este ión (Fig 6B).
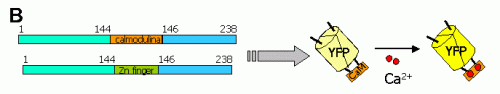 |
Figura 6B: En esta posición se toleran inserciones mayores, como CaM o un dedo de zinc. |
En las permutaciones circulares, los extremos N-t y C-t de GFP se unen por un hexapéptido (GGTGGS) (Fig 6C). La Tyr145 se sustituye por una Met que es ahora el primer aminoácido, y el residuo 144 original pasa a ser el último de la quimera. Sobre esta GFP permutada circularmente (cpGFP) se introdujeron fusiones al principio (péptido M13) y al final (CaM) con lo que se crearon nuevos sensores de Ca 2+ llamados pericams (Fig 6D), que cambian sus propiedades fluorescentes de forma reversible con un aumento del ión Ca 2+ : aumentan la emisión ( flash-pericam ), se apagan ( inverse-pericam ), o cambian su pico de excitación de 415 nm a 494 nm ( ratiometric pericam ) 29,30 . Se ha descrito la generación de ratones transgénicos con dos indicadores de Ca 2+ funcionales, inverse pericam y camgaroo -2, bajo el control de un promotor inducible por tetraciclina 31 , lo que permitirá realizar estudios de activación de poblaciones celulares, por ejemplo en rodajas de cerebro, utilizando los cambios rápidos de Ca 2+ como un indicador de actividad neuronal.
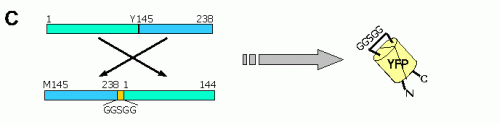 |
Figura 6C: La YFP permutada circularmente retiene fluorescencia |
 |
Figura 6D: Fusiones de CaM y péptido M13 constituyen una familia de indicadores de Ca 2+ llamados “pericams”. |
Una aplicación que empieza a dar frutos, aunque tiene limitaciones, es la utilización de FRET para detectar interacciones entre proteínas 32 . Como muestra el esquema de la figura 7, usando como ejemplo el receptor de ácido retinoico (RAR) y un fragmento del coactivador SRC-1 33 , se trata de marcar dos proteínas cuya interacción se quiere medir con sendas FPs, por ejemplo ECFP y EYFP. Se expresan las proteínas de fusión en las células, y si existe interacción entre las proteínas marcadas, aparecerá una señal FRET, siempre que se den las condiciones favorables de distancia y orientación entre las FPs a las que nos hemos referido antes.
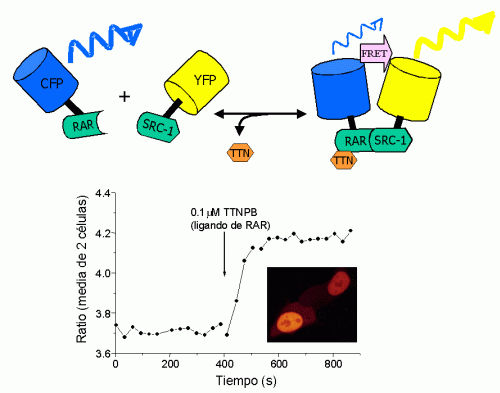 |
Figura 7: FRET intermolecular para cuantificar interacciones entre proteínas. Se cotransfectaron fusiones de CFP con el receptor de ácido retinoico (RAR) y de YFP con un fragmento del coactivador SRC-1, que se expresan en el núcleo celular. La interacción entre ambas proteínas quiméricas en respuesta a un ligando del receptor (TTNPB) induce una señal FRET (trazo) cuantificable con buena resolución temporal. |
Como limitaciones de esta técnica mencionaremos que las proteínas quiméricas deben mantener la función de las proteínas nativas; es necesario transferir cDNA (o mRNA) mediante transfección, transducción viral o microinyección y esperar a que se sinteticen las proteínas; es dificil de ajustar la concentración relativa de ambas proteínas, que debería ser igual para una óptima detección de FRET (esta relación siempre se mantiene constante en los camaleones); el cociente señal/ruido está limitado por la mala separación espectral entre ECFP y EYFP, aunque esperamos que esto mejore en el futuro con el uso de nuevas variantes de FPs rojas. Además, se requieren controles negativos de interacción; el mejor control sería introducir una mutación que impida la interacción entre las proteínas. En la determinación de FRET intermolecular con frecuencia aparecen falsos negativos, probablemente porque la distancia entre las FPs en el complejo de las proteínas que interaccionan es mayor a la que permite FRET (aproximadamente de 3 a 10 nm para ECFP/EYFP), o porque su orientación en el complejo es desfavorable. También hay que considerar que la presencia de proteínas endógenas (no marcadas con FPs) puede disminuir la sensibilidad del método, ya que en ese caso pueden formarse complejos de proteína marcada y no marcada, los cuales no producen señal FRET.
Pese a todas estas reservas, FRET presenta ventajas frente a otras técnicas de detección de interacciones moleculares. Por ejemplo, la interacción puede estudiarse en el interior de cualquier célula y no está limitada al núcleo de la levadura, como ocurre con la técnica del doble híbrido. Cuando se demuestra FRET intermolecular fehacientemente, la información obtenida supera en resolución temporal y espacial a la obtenida con cualquier otra técnica.
Nosotros recomendamos que se use un control positivo cuando un investigador se enfrenta a un proyecto de FRET por vez primera, para cerciorarse de que todos los elementos físicos del equipo y del programa informático se han seleccionado correctamente. Por ejemplo, se puede usar un camaleón en presencia de Ca 2+ . También hemos construido proteínas de fusión con ECFP y EYFP que presentan una alta eficiencia de FRET (50-60%) y que ponemos a disposición de los laboratorios que las soliciten.
Se han descrito al menos cuatro estrategias para cuantificar FRET. A) S e puede medir el acortamiento de la vida media del estado excitado del donante mediante FLIM o fluorescence lifetime imaging 32 , aunque esto requiere tecnología compleja. B) También se puede medir la disminución de la emisión del donante ( quenching ) y el aumento de la emisión del aceptor (emisión sensibilizada), ambas mientras se excita sólo el donante con luz de 430 nm (o mediante un láser de 458 nm en un microscopio confocal). El ratio de las imágenes aceptor/donante mejora el cociente señal/ruido y es proporcional a la tasa de FRET 34 . C) Otro método se basa en la característica diagnóstica de FRET de que aumenta la emisión del donante si se destruye el aceptor (por ejemplo, mediante “fotobleaching”) y permite calcular la eficiencia absoluta de FRET 34 . D) Por último, se pueden separar espectralmente las imágenes del donante de energía (CFP) y de emisión sensibilizada del aceptor (YFP) mediante algoritmos que usan la contribución de cada proteína a los dos canales de detección 35,36 . El ratio de la imagen de emisión sensibilizada (YFP FRET ) y la imagen de YFP excitada en su máximo de absorción (YFP referencia ), ambas corregidas espectralmente, es una buena forma de expresar la tasa de FRET.
La contribución de las proteínas fluorescentes a nuestra comprensión de la dinámica celular y las redes de interacción molecular no ha hecho sino empezar. El progreso en tecnología microscópica al que asistimos (confocal, multifotón, cámaras CCD con mayor sensibilidad, etc.), así como la generación de FPs mejoradas y de distintos colores 37,38,39 , permitirá sin duda la construcción de sensores para una mayor variedad de procesos bioquímicos y su estudio a nivel subcelular, celular y en organismos completos 40,31 .
FRET, transferencia de energía resonante de fluorescencia; FP, del inglés fluorescent protein o proteína fluorescente; GFP, proteína fluorescente verde; CFP, proteína fluorescente cian (verde azulada); YFP, proteína fluorescente amarilla; RFP, proteína fluorescente roja; CaM, calmodulina; R.C., rendimiento cuántico.