La autofagia es un proceso catabólico conservado en todas las células eucariotas, que implica la degradación del material citoplasmático en la vacuola/lisosoma 1,2 . En la levadura Saccharomyces cerevisiae la autofagia se induce bajo condiciones de ayuno y permite a la célula sobrevivir períodos prolongados de escasez de nutrientes al reciclar productos de su propia degradación 2 . El transporte del material citoplasmático hasta la vacuola se produce a través de unas vesículas de doble membrana, los autofagosomas, que durante su formación atrapan porciones de citosol de las inmediaciones 3 . La membrana externa del autofagosoma se fusiona con la membrana vacuolar liberando en su lumen un cuerpo autofágico, una vesícula de contenido citoplasmático delimitada por una única membrana, que finalmente es degradado por las hidrolasas residentes en la vacuola 2,3 (Figura 1) .
 |
Figura 1: Transporte y maduración de Ape1 a través de las rutas cvt y autofagia. Ape1 es sintetizada en el citosol en forma de precursor inactivo (prApe1). Después de su transporte a la vacuola, a través de los autofagosomas o de las vesículas cvt, se activa por proteolisis generándose la forma madura (mApe1) de la proteína. Adicionalmente, los autofagosomas, de mayor tamaño que las vesículas cvt, incorporan de modo no selectivo el citosol de las inmediaciones y lo transportan hasta la vacuola para su degradación. CIT, citoplasma; VAC, vacuola; PAS, estructura preautofagosomal. |
A principios de la década de los noventa dos laboratorios aislaron de forma independiente una serie de mutantes de S. cerevisiae defectivos en autofagia, los mutantes aut y apg 4,5 . Hasta ese momento la mayor parte de los conocimientos sobre autofagia eran exclusivamente morfológicos, sin embargo, con la identificación y caracterización de los genes APG / AUT se han podido establecer las bases moleculares del proceso autofágico tomando como modelo eucariótico a la levadura S. cerevisiae 6 Tras la clonación de estos genes se comprobó que la mayoría de ellos eran alélicos con los genes CVT ( C ytosol to V acuole T argeting), implicados en la ruta cvt de transporte de las hidrolasas aminopeptidasa 1 (Ape1) y a-manosidasa (Ams1) desde el citosol hasta la vacuola 7 . La gran cantidad de genes compartidos por la autofagia y la ruta cvt se refleja en que la morfología y los mecanismos moleculares de ambos procesos son similares 8,9 Aun así existen claras diferencias entre las dos rutas; la autofagia es un proceso catabólico e inducible en el que los autofagosomas, con un tamaño de 300-900 nm de diámetro, transportan masivamente porciones de citoplasma y otros orgánulos hasta la vacuola. Por el contrario, la ruta cvt es un proceso biosintético y constitutivo en el que las vesículas cvt, de 140-150 nm de diámetro, transportan específicamente Ape1 y Ams1. Los autofagosomas, al igual que las vesículas cvt, también incorporan selectivamente Ape1 y Ams1 como parte de la carga (Figura 1).
Con el objetivo de unificar la nomenclatura de los genes APG , AUT y CVT recientemente se ha propuesto renombrar todos ellos como genes ATG; genes relacionados con la A u T ofa G ia 10 . Hasta la fecha se han identificado 25 genes ATG implicados en la autofagia y/o ruta cvt, 16 de los cuales resultan esenciales en el proceso de formación de los autofagosomas. 17 genes ATG son compartidos por la autofagia y la ruta cvt, 7 son específicos de la ruta cvt pero no de la autofagia, y 1 es específico del proceso autofágico y no de la ruta cvt. Una descripción de todos ellos se presenta en la tabla 1. Además de los genes ATG se han identificado otros genes esenciales en la autofagia, involucrados en eventos de fusión de las vesículas a nivel vacuolar, que son comunes a otras rutas de transporte vesicular 11
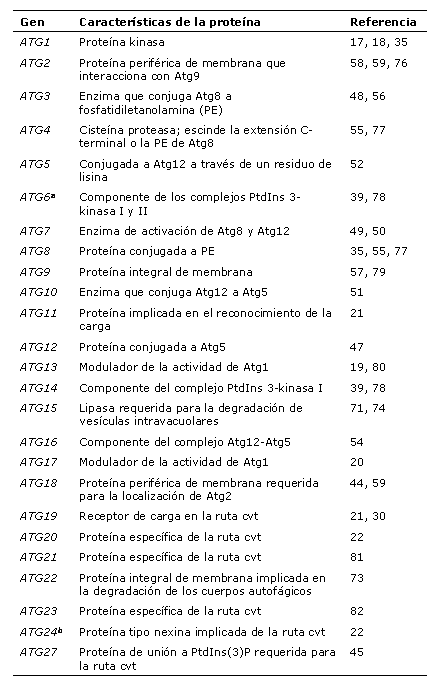 |
Tabla 1 . Descripción de los genes relacionados con la autofagia. a El nombre estándar de este gen es VPS30 . b El nombre estándar de este gen es SNX4 . |
A pesar de los conocimientos adquiridos durante una década de estudios moleculares en el campo de la autofagia, no se ha identificado el origen de la membrana empleada por las células de levadura para la formación de los autofagosomas. Recientemente se ha identificado una región puntual adyacente a la vacuola donde localizan in vivo la mayor parte de las proteínas Atg 12,13 . Se postula que en esta región, denominada estructura pre-autofagosomal (PAS de “ P re- A utophagosomal S tructure”), podría estar el origen de la membrana de los autofagosomas 14 .
Para facilitar la comprensión del proceso autofágico, éste puede desglosarse en varias etapas; (1) inducción, (2) selección de la carga de transporte, (3) formación de los autofagosomas, (4) reciclaje de proteínas Atg, (5) fusión de los autofagosomas con la vacuola y (6) hidrólisis final de la carga en el lumen vacuolar. A continuación se pasa a describir cada una de las etapas por separado revisando los mecanismos moleculares implicados en cada una de ellas.
La inducción de la autofagia no requiere de la síntesis de novo de proteínas e implica un mecanismo de transducción de señales dependiente de la actividad de la proteína Tor 15 . Tor es una serina treonina kinasa, perteneciente a la familia de proteínas relacionadas con las fosfatidil inositol kinasas, cuya actividad es sensible a la disponibilidad de nutrientes en el medio, actuando como un integrador de las respuestas celulares frente al ayuno en eucariotas. En condiciones de crecimiento Tor es activo y la autofagia se encuentra inhibida. Por el contrario, situaciones de ayuno provocan una inactivación de Tor y la concomitante inducción del proceso autofágico 16 .
Se desconoce como Tor transmite esta señal de ayuno a la maquinaria Atg para que tenga lugar el desarrollo de la autofagia. Una hipótesis es que se produce a través de la modulación de la actividad kinasa de Atg1, una serina treonina kinasa esencial tanto para la ruta cvt como para la autofagia 17,18 . Se ha demostrado que Atg1 interacciona con Atg13 de un modo dependiente a la actividad de Tor 19,20 . Adicionalmente, Atg1 es capaz de interaccionar con otras proteínas Atg que formarían parte de un teórico complejo Atg1 kinasa dinámico sensible a la escasez de nutrientes en el medio externo. Entre estas proteínas se encuentran Atg11, específica de la ruta cvt, y Atg17, específica de la autofagia 20,21 Adicionalmente, Atg13 es capaz de interaccionar con Vac8, mientras que Atg17 interacciona con las proteínas específicas de la ruta cvt Atg20 y Atg24 22
Kabeya et al . (2005) 23 han propuesto un modelo para el mecanismo de transducción de señales que conducen a la inducción de la autofagia. Bajo condiciones de abundancia nutricional Atg13 se encuentra hiperfosforilada de un modo dependiente de la actividad de Tor 20 Tras la inhibición de Tor como consecuencia de un estímulo causado por el ayuno, Atg13 resulta parcialmente defosforilado favoreciéndose su interacción con Atg17 23 El complejo Atg13-Atg17 se asocia con Atg1, lo que provoca una inducción de su actividad kinasa, que a su vez desencadena finalmente la formación de los autofagosomas 20,23 .
Sin embargo, la implicación de la actividad kinasa de Atg1 en la autofagia es un punto controvertido. Estudios previos de Matsuura et al . (1997) 17 demostraron que la inducción de la autofagia implica una reducción de la actividad kinasa de Atg1, medida como actividad autofosforilante. Más recientemente, Abeliovich et al . (2003) 24 determinaron que la inhibición de la actividad kinasa de Atg1 no afectaba al desarrollo normal de la autofagia aunque bloqueaba la ruta cvt. Construyeron una cepa mutante en ATG1 con una mutación puntual en el dominio kinasa de la proteína que la hacía específicamente sensible a la inhibición por el compuesto 1-NA-PP1 (derivado del inhibidor general de kinasas PP1). Bajo estas condiciones de inhibición de la actividad kinasa de Atg1 se conseguía un bloqueo del transporte de la Ape1 a través de la ruta cvt, sin que tuviera ningún efecto sobre el transporte a través de la ruta autofágica. Según este resultado, la actividad kinasa de Atg1 sería esencial para el funcionamiento de la ruta cvt, mientras que para el desarrollo del proceso autofágico, Atg1 tendría un papel fundamentalmente estructural e independiente de su actividad kinasa.
Adicionalmente, Atg1 parece estar implicado en etapas posteriores a la inducción de la autofagia, como son la selección de la carga, tal y como revela su interacción con Atg11 (ver más adelante), y el reciclaje de proteínas Atg durante la formación de autofagosomas, para asegurar la continuidad del proceso 25 .
Durante la autofagia el citoplasma es incluido en los autofagosomas de un modo no selectivo. Sin embargo, existen proteínas cuya incorporación se ve favorecida con respecto al resto de constituyentes de la célula y son transportadas selectivamente hasta la vacuola durante la autofagia. Entre estas proteínas se encuentran la Ape1 y la Ams1. Recientemente se ha demostrado que la acetaldehído deshidrogenasa (Ald6) también es incorporada selectivamente en los autofagosomas durante el proceso autofágico 26 .
Ape1, sintetizada en el citosol como un precursor inactivo (prApe1), rápidamente oligomeriza formando homo-dodecámeros que se organizan en una estructura de orden superior, el “complejo Ape1” 27,28 . La proteína citosólica Atg19 actúa como un receptor de Ape1 y Ams1, pudiendo interaccionar simultáneamente con el propéptido de la prApe1 del “complejo Ape1” así como con la proteína Ams1 29,30 . Esta estructura proteica constituida por prApe1, Ams1 y Atg19 es conocida con el nombre de complejo cvt 28 (Figura 2) . La localización del complejo cvt en la PAS depende de su interacción con la proteína Atg11 28 . Atg11 actúa como una molécula adaptadora permitiendo el anclaje del complejo cvt a la membrana de la PAS, conectando de esta forma la maquinaria implicada en la formación de la vesícula con la carga a transportar 31 . La ubicación del complejo carga-receptor en la PAS desencadena la organización de la maquinaria molecular implicada en la formación de la vesícula cvt y en consecuencia la formación de la vesícula en torno al complejo cvt 31,32 . Sin embargo, la localización del complejo cvt en la PAS no parece afectar a la organización de la PAS durante la autofagia, al proceso de formación de los autofagosomas o al desarrollo normal de la autofagia no selectiva 31,33 .
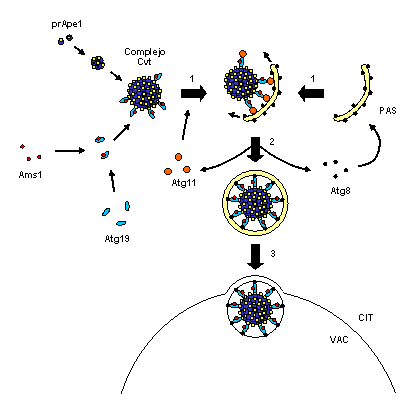 |
Figura 2: Proceso de selección de carga durante la ruta de transporte cvt. (1) El complejo cvt es anclado a la membrana de la PAS a través de la proteína adaptadora Atg11, que interacciona directamente con Atg19, el receptor de Ape1 y Ams1. (2) Expansión de la membrana de la PAS hasta formar una vesícula cvt que contiene el complejo cvt en su interior. (3) La membrana externa de la vesícula cvt se fusiona con la membrana de la vacuola, liberando un cuerpo cvt al lumen vacuolar. prApe1, precursor citosólico inactivo de la aminopeptidasa 1; Ams1, α-manosidasa 1; CIT, citoplasma; VAC, vacuola; PAS, estructura preautofagosomal. |
Si bien los mutantes defectivos en ATG11 son incapaces de transportar prApe1 a la vacuola a través de la ruta cvt, se ha demostrado que estos mutantes desarrollan un proceso autofágico normal, en el que el complejo cvt es incorporado correctamente en los autofagosomas 29 Esto significa que debe existir otra molécula funcionalmente análoga a Atg11 implicada en el anclaje del complejo cvt en la PAS bajo condiciones de ayuno. Un candidato para actuar como molécula adaptadora durante la autofagia es la proteína Atg8, que junto con su localización en la PAS se ha demostrado interaccionar con el receptor Atg19 13,28 .
La prApe1 constituye un buen marcador molecular de las vesículas cvt y de los autofagosomas. Estos intermedios de transporte se acumulan en el citoplasma de los mutantes ypt7 que son incapaces de llevar a cabo eventos de fusión de membranas que implican a la vacuola 34 . En un fondo genético ypt7 , la proteinasa K no es capaz de hidrolizar la prApe1 cuando se añade sobre esferoplastos. Esto es debido a que la prApe1 queda protegida de la acción de la hidrolasa a través de las membranas de las vesículas cvt y de los autofagosomas acumulados. Por el contrario, en los mutantes defectivos en la formación de estas vesículas, la prApe1 ya no queda protegida por las membranas de las vesículas, siendo ahora accesible a la actividad hidrolítica de la proteinasa K, lo que se observa como un cambio en la movilidad electroforética de la proteína hasta su forma madura 1,35,36 . Este ensayo bioquímico permite distinguir los genes autofágicos implicados en la formación de los autofagosomas de aquellos que intervienen en etapas posteriores del proceso, como la fusión con la vacuola o la degradación de los cuerpos autofágicos.
La mayor parte de los genes ATG identificados hasta la fecha están directamente implicados en la formación de los autofagosomas. Sin embargo, los mutantes atg8 desarrollan unas estructuras con un contenido análogo al de los autofagosomas, aunque de menor tamaño, que suelen aparecer también en menor número y en ocasiones con formas aberrantes 15,36 . Estas estructuras, sin ser plenamente funcionales, son capaces de transportar algo de Ape1 a la vacuola en condiciones de ayuno así como soportar una autofagia reducida que confiere una mayor resistencia frente al ayuno con respecto a otros mutantes atg . Este mismo fenotipo se ha observado en células ayunadas en presencia de cicloheximida, un inhibidor de la síntesis de proteínas 15 . Atg8 es la única proteína conocida implicada directamente en el proceso autofágico cuya expresión se induce bajo las condiciones que desencadenan el proceso autofágico 36 . Como se mencionó anteriormente, el desarrollo de la autofagia es independiente de la síntesis de novo de proteínas, aunque para que los autofagosomas adquieran un tamaño adecuado es necesaria la transcripción y traducción de nuevos genes, entre los que se encuentran ATG8 .
Estas evidencias fenotípicas indican que los mutantes atg8 no son completamente defectivos en autofagia y se postula que Atg8 podría estar implicado en la expansión de los autofagosomas, contribuyendo a la formación de vesículas con un tamaño funcional. Así pues, el fenotipo de los mutantes atg8 permite diferenciar las etapas de nucleación y expansión en el proceso de formación de los autofagosomas 15 .
La nucleación implica el inicio de la formación de los autofagosomas con la reunión de todos los componentes necesarios en la PAS; las membranas que constituirán los límites de los autofagosomas, la carga que quedará finalmente incluida en ellos así como el conjunto de proteínas Atg requeridas para la formación de las vesículas autofágicas. Cabe destacar que Atg8 es la única proteína autofágica conocida que queda incluida en los autofagosomas maduros, lo que conlleva finalmente su transporte hacia la vacuola durante la autofagia. Esto ha permitido utilizar a Atg8 como marcador de la ruta autofágica y seguir morfológicamente el proceso identificándose estructuras intermedias del proceso de formación de los autofagosomas 36,37,38 .
Las proteínas Atg implicadas en la formación de los autofagosomas pueden clasificarse en diferentes grupos funcionales. La mayor parte de ellas forman parte de un sistema de conjugación de proteínas esencial para la autofagia que se asemeja al proceso de ubiquitinación de proteínas. Otro grupo de proteínas Atg es integrante de un complejo fosfatidilinositol 3-kinasa (PtdIns 3-kinasa). A continuación se describen los mecanismos moleculares implicados en el proceso de formación de los autofagosomas.
Vps34, la única PtdIns 3-kinasa de S. cerevisiae , puede formar parte de dos complejos diferentes implicados en procesos de transporte vesicular 13,22,39 . El complejo I, integrado por las proteínas Vps34, Vps15, Vps30/Atg6 y Atg14, es esencial en el desarrollo de las rutas cvt y autofagia, mientras que el complejo II, con la proteína Vps38 sustituyendo a Atg14, es requerido en la ruta vps ( V acuolar P rotein S orting) implicada en el transporte de la hidrolasa vacuolar carboxipeptidasa Y (CPY) desde el Golgi hasta la vacuola 39,40,41 .
Se desconoce cual es la función que desempeña el complejo I PtdIns 3-kinasa en el proceso autofágico. Una posibilidad es que el PtdIns(3)P sintetizado por este complejo se localice específicamente en la membrana de la PAS permitiendo el reclutamiento de proteínas Atg en esta estructura, y con ello la función concertada de las proteínas para la formación de los autofagosomas. De hecho, una fracción de Atg14 se ubica en la PAS y varias proteínas Atg de unión a PtdIns(3)P, cuya localización en la PAS depende de la función del complejo I PtdIns 3-kinasa, interaccionan con otras proteínas Atg permitiendo su asociación con la PAS. Entre estas proteínas de unión a PtdIns(3)P se encuentran Mai1/Atg21, Atg18, Atg20, Atg24 y Etf1/Atg27. Mai1/Atg21 es una proteína esencial para la ruta cvt pero no para la autofagia y está implicada en la lipidación de Atg8 así como en su localización en la PAS 42,43 . De modo similar, la proteína Atg18, requerida en las rutas cvt y autofagia, es necesaria para la correcta localización de Atg2 en la PAS 44 . Atg20 y Atg24, esenciales para la ruta cvt pero prescindibles en el transporte autofágico, interaccionan con Atg17 y por lo tanto podrían formar parte del teórico complejo Atg1 kinasa implicado en la señalización de la autofagia 22 . Etf1/Atg27 es otra de las proteínas de unión a fosfoinosítidos esencial para la ruta cvt pero no para la autofagia 45 .
El proceso de formación de autofagosomas requiere de dos complejos de conjugación cuyo mecanismo de síntesis es molecularmente similar al proceso de ubiquitinación de proteínas 46 . Uno de estos complejos es el formado por el conjugado Atg12-Atg5 47 mientras que el otro implica la unión covalente de Atg8 con una molécula de fosfatidiletanolamina (Atg8-PE) 48 (Figura 3).
La formación del complejo Atg12-Atg5 comienza con la activación inicial de Atg12 49 mediante la formación de un enlace tioéster de alta energía entre la cisteína 507 de Atg7 y la glicina 186 C-terminal de Atg12 47,50 (Figura 3A (1)). Posteriormente, Atg12 es transferido a la cisteína 133 de Atg10 51 que finalmente cataliza la formación del conjugado Atg12-Atg5 a través de un enlace isopeptídico entre la glicina 186 de Atg12 y la lisina 149 de Atg5 47,52 (Figura 3A (2)). El conjugado Atg12-Atg5 interacciona con Atg16 formando un complejo multimérico de 350 kDa esencial para el desarrollo de la autofagia 53,54 (Figura 3A (3)).
|
Figura 3:Sistemas de conjugación requeridos para el desarrollo de la autofagia en la levadura S. cerevisiae . A. Formación del complejo Atg12-Atg5. (1) Activación de la proteína Atg12. (2) Formación del conjugado Atg12-Atg5. (3) Oligomerización del conjugado Atg12-Atg5 con Atg16 en un complejo de 350 kDa. B. Mecanismos moleculares implicados en la formación del complejo Atg8-PE. (1) Eliminación del residuo de arginina C-terminal de Atg8. (2) Activación de la proteína Atg8. (3) Formación del conjugado Atg8-PE. (4) Localización del conjugado Atg8-PE en la estructura preautofagosomal. (5) Liberación de Atg8 de la estructura preautofagosomal. Algunos aminoácidos importantes involucrados directamente en el mecanismo de conjugación son representados en determinadas proteínas (G, Glicina; K, Lisina; R; Arginina). El asterisco sobre las proteínas simboliza un estado activado de las mismas mediante la formación de un enlace covalente de alta energía con Atg7. PE, fosfatidiletanolamina; PAS, estructura preautofagosomal. |
El sistema de conjugación de Atg8 implica la escisión inicial de la arginina 117 C-terminal de Atg8 por medio de la cisteína proteasa Atg4 55 (Figura 3B (1)). A través de la glicina 116 que constituye el nuevo extremo C-terminal de Atg8, y de modo análogo al mecanismo de activación de Atg12, se forma un enlace tioéster de alta energía con Atg7 (Figura 3B (2)). Atg8 es entonces transferido a Atg3, que se encarga de catalizar la formación del conjugado Atg8-PE 38,56 (Figura 3B (3)). Posteriormente, el complejo Atg8-PE, de un modo dependiente de los complejos PstIns 3-kinasa I y Atg12-Atg5 y las proteínas Atg9 y Mae1/Atg21, se localiza en la membrana de la PAS 38 (Fig. 3B (4)). Atg8-PE es un complejo dinámico, resultando necesaria la escisión del conjugado de la PAS por parte de Atg4 para el correcto funcionamiento de la autofagia 55 (Fig. 3B (5)). El Atg8 liberado sería susceptible para entrar en nuevas rondas de conjugación.
Atg9 es una proteína transmembrana esencial para la autofagia y la ruta cvt 57 Además de localizarse en la PAS, aparece distribuida en regiones puntuales dispersas por el citoplasma, que no se corresponden con ninguno de los orgánulos que componen el sistema endomembrana, las vesículas cvt o los autofagosomas 25,57 . Pese a su condición de proteína transmembrana, Atg9 queda excluida de los autofagosomas, al menos antes de su fusión con la vacuola, habiéndose observado un transporte retrógrado de esta proteína desde la PAS a las regiones puntuales citoplasmáticas, posibles centros donadores de membrana para la construcción de autofagosomas desde la PAS 25 . Este fenómeno de transporte retrógrado resulta esencial para la autofagia. Para dicho transporte son necesarias proteínas autofágicas adicionales como Atg2 58,59 , Atg18 60 , el complejo I PtdIns 3-kinasa 39 y la función kinasa independiente de Atg1 junto con Atg13 25 . De acuerdo con el modelo planteado por Reggiori et al (2004) 25 , el PtdIns-3-fosfato (PtdIns(3)P) generado en la PAS por el complejo PtdIns 3-kinasa I desencadenaría el reclutamiento secuencial de proteínas Atg imprescindible para el transporte retrógrado de Atg9. El complejo Atg1-Atg13 induciría la asociación de Atg2 y Atg18, proteína de unión a PtdIns(3)P, con Atg9 una vez concluída la función de esta proteína en la PAS. La formación de este complejo iniciaría el reciclaje de Atg9 desde la PAS.
Atg2 es una proteína esencial para las rutas cvt y Atg que se distribuye por el citoplasma y la PAS de la célula. Su localización en la PAS es dependiente de Atg9, proteína con la que interacciona físicamente, teniendo en esta región un comportamiento bioquímico típico de una proteína periférica de membrana 59 .
Las células eucariotas utilizan un sistema de vesículas para transportar proteínas entre los orgánulos que componen el sistema de endomembranas. Para que una proteína alcance el orgánulo donde realiza su función es necesario un reconocimiento molecular específico entre el orgánulo aceptor y la vesícula de transporte que porta la carga proteica. En estos procesos de fusión están implicadas proteínas como NSF/Sec18 y a-SNAP/Sec17 61 , proteínas SNARE de la membrana del orgánulo aceptor (t-SNARE) y de la vesícula de transporte (v-SNARE) 62,63 , y el complejo HOPS/VPS de clase C 64 . El requerimiento de estas proteínas es una constante en los procesos de fusión de membranas, y la fusión de los autofagosomas con la vacuola no es una excepción.
Vam3 es una t-SNARE de la membrana vacuolar esencial para las rutas cvt y autofagia, habiéndose observado autofagosomas en el citoplasma de células defectuosas en la función de Vam3 65,66 Por otro lado la v-SNARE Vti1 también es esencial para la fusión de los autofagosomas con la vacuola 67 . Un fenotipo similar se ha observado en mutantes ccz1 68 . Ccz1 se ha demostrado que actúa junto con Mon1 en los procesos de fusión vacuolar 69
Al igual que la Ape1, Atg8 se ha utilizado como marcador molecular de los autofagosomas. El lumen de los autofagosomas maduros presenta Atg8, apareciendo también asociado a la superficie luminal de la membrana vesicular. Trabajos de inmunomicroscopía electrónica han revelado estructuras membranosas, posibles presursores de los autofagosomas, con un elevado contenido en Atg8. Igualmente, los autofagosomas inmaduros presentan Atg8 asociado con su superficie externa, a diferencia de las vesículas maduras. En este sentido Atg8 podría formar parte de una posible cubierta proteica en los autofagosomas, cuya desorganización permitiría la fusión de los autofagosomas, de un modo análogo a lo que ocurre con las cubiertas proteicas de las vesículas COPII 70 .
A diferencia de las rutas clásicas de tráfico de proteínas, la autafagia utiliza vesículas de doble membrana como intermedios de transporte. Esto implica que tras la fusión del autofagosoma con la vacuola, su contenido citoplasmático no es liberado directamente al lumen vacuolar sino que queda delimitado por una membrana constituyendo el cuerpo autofágico. Para que las hidrolasas vacuolares puedan acceder al citoplasma transportado es necesaria la degradación previa de la membrana del cuerpo autofágico. Se han identificado dos proteínas Atg vacuolares esenciales para el proceso de degradación de los cuerpos autofágicos; Atg15, una lipasa transportada a la vacuola a través de la ruta MVB (del inglés M ulti V esicular B odies) y Atg22, una posible permeasa de la membrana vacuolar 71,72,73,74 . Defectos funcionales en estas proteínas causan la acumulación de cuerpos autofágicos en la vacuola así como una reducción en la actividad hidrolítica celular en condiciones que inducen la autofagia 71,73 . Junto con estas proteínas Atg, la actividad de varias proteinasas vacuolares así como la acidificación vacuolar son factores esenciales para la degradación de los cuerpos autofágicos 2,75 .
Resumiendo, aunque la autofagia ha sido objeto de investigación durante más de cincuenta años, la elección de S. cerevisiae como modelo eucariótico para su estudio ha proporcionado, en poco más de de diez años, una gran cantidad de información sobre los eventos moleculares que rigen el proceso autofágico. Aunque todavía importantes preguntas permanecen sin respuesta, por ejemplo aun no se sabe con certeza el origen de la membrana autofagosomal, la aplicación de la bioquímica, la genética molecular y de sofisticadas técnicas microscópicas resolverán estos y otros problemas relacionados con la autofagia. Adicionalmente, muchos de los genes implicados en autofagia identificados en S. cerevisiae tienen sus respectivos homólogos en organismos eucariotas superiores, lo que ha facilitado el estudio de la autofagia en otros sistemas más complejos, lo que ha abierto nuevas líneas de investigación en este campo, como puede ser la fisiología celular y el papel de la autofagia en enfermedades humanas.